Capítulo 5 Enzimas
R.De modo geral, a equação simplificada que descreve a atividade de uma enzima E sobre um substrato S pode ser descrita como:
Onde P representa o produto da reação, ES o complexo ativado no estado de transição, e k1, k2 e k3 as constantes de velocidade da reação.
Onde Km representa a constante de Michaelis-Menten, e Vm a velocidade limite da reação (por vezes denominada erroneamente como velocidade máxima, embora a hipérbole quadrática descrita pela função não exiba valor máximo por não atingir uma assíntota). Por sua vez Km pode ser definido a partir das constantes de velocidade da equação (5.1) como:
\[\begin{equation} Km=\frac{k1+k3}{k2} \tag{5.2} \end{equation}\]Vm= 10
Km = 0.5
curve(Vm*x/(Km+x),xlim=c(0,10),
xlab="[S]", ylab="v")
abline(h=5,lty=2,col="blue")
abline(v=0.5,lty=2, col="blue")
text(x=1,y=0.2,"Km",col="blue")
text(1,5.3,"Vm/2",col="blue")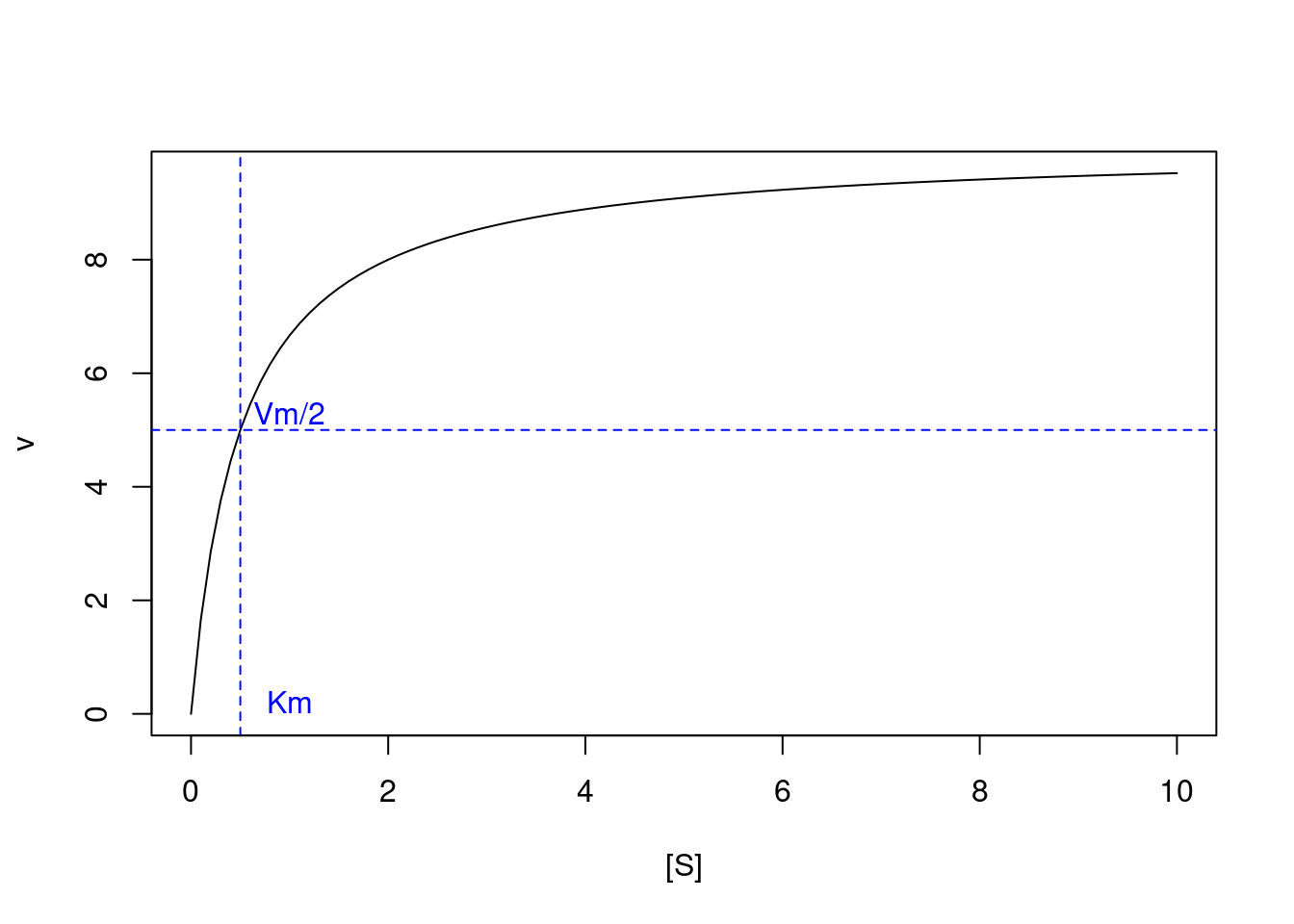
Figura 5.1: Curva de Michaelis-Mentem para uma enzima exibindo Vm=10 e Km=0,5 (50 curvas).
Vm=10
Km=seq(from=0.1,to=10,by=0.2) # sequência para 50 valores de Km
for(i in 1:length(Km)) { # loop para adicionar curva de Michaelis-Mentem a cada valor de Km
add<-if(i == 1) FALSE else TRUE # controle de fluxo que permite adição de curva a partir da segunda iteração (ou seja, quando i > 1)
curve(Vm*x/(Km[i]+x), col= i, lwd=0.8, from =0, to=10, n =100,
xlab="[S}", ylab="v", add=add)
}
arrows(0.5,9,3,6,length=0.1,angle=45, col="blue") # seta para Km
text(0.2,9,"Km",col="blue") # indexador para Km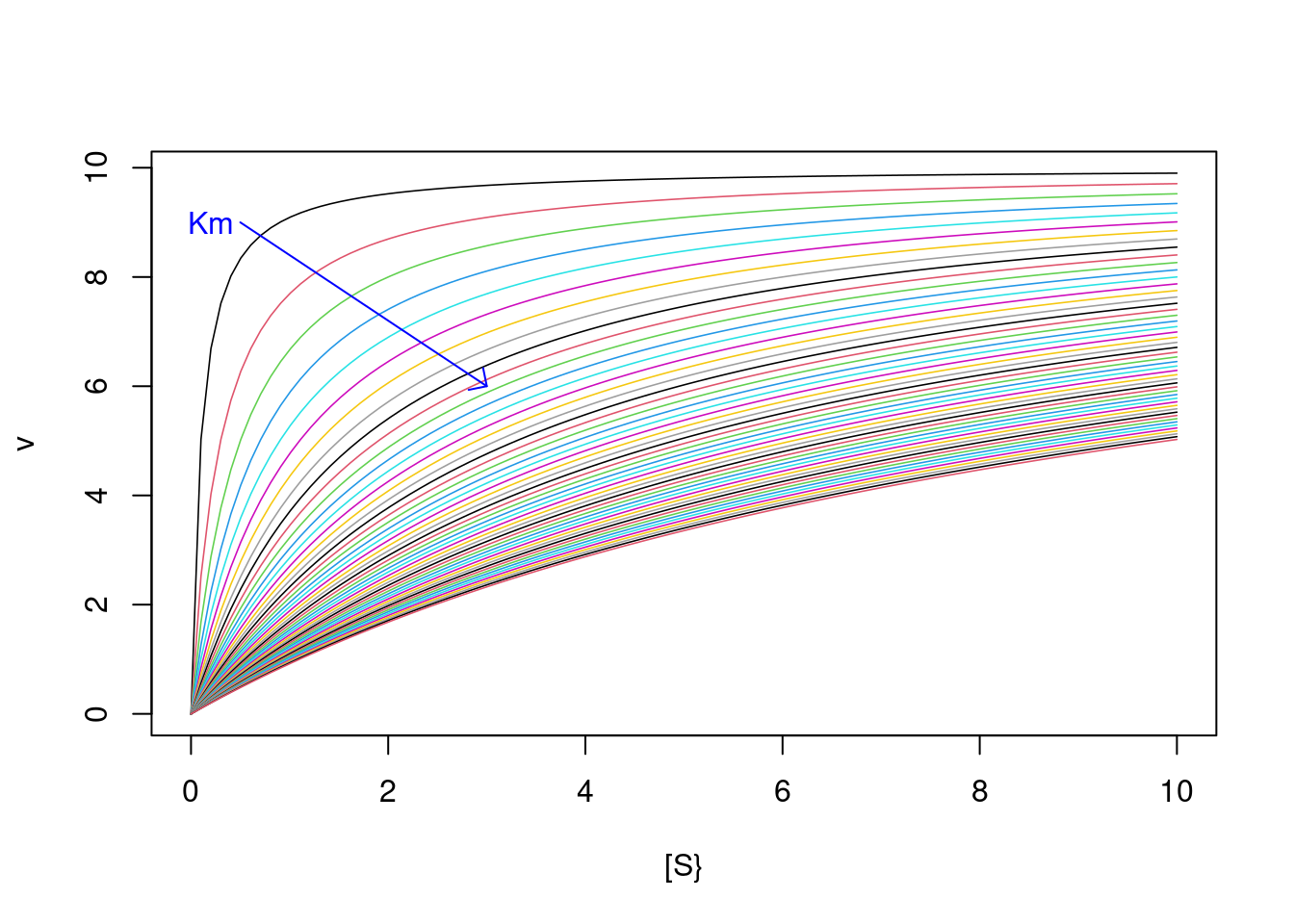
Figura 5.2: Curvas de Michaelis-Menten com variação iterativa para Km de 0.1 a 10.
Não obstante, a equação que descreve a equação de Michaelis-Mentem constitui uma hipérbole quadrática e, como tal, não possui assíntota matemática, apenas visual. De fato, os próprios autores do trabalho original, Leonor Michaelis e Maud Mentem, reportaram seus dados com a representação de S em eixo logaritmo permitindo melhor visualização da região assintótica do gráfico (Michaelis and Menten 1913).
5.1 Obtenção de parâmetros cinéticos a partir de dados expermentais simulados
Vm=10
Km=0.5
set.seed(1500) # fixa a semente para geração de dados aleatórios reproduzíveis
erro=runif(20,0,1) # comando para erro uniforme (no. de pontos, min, max)
curve(Vm*x/(Km+x)+erro, type="p", from =0, to=1, n =20,
xlab="[S}", ylab="v") # elaboração da curva com cômputo de erro uniforme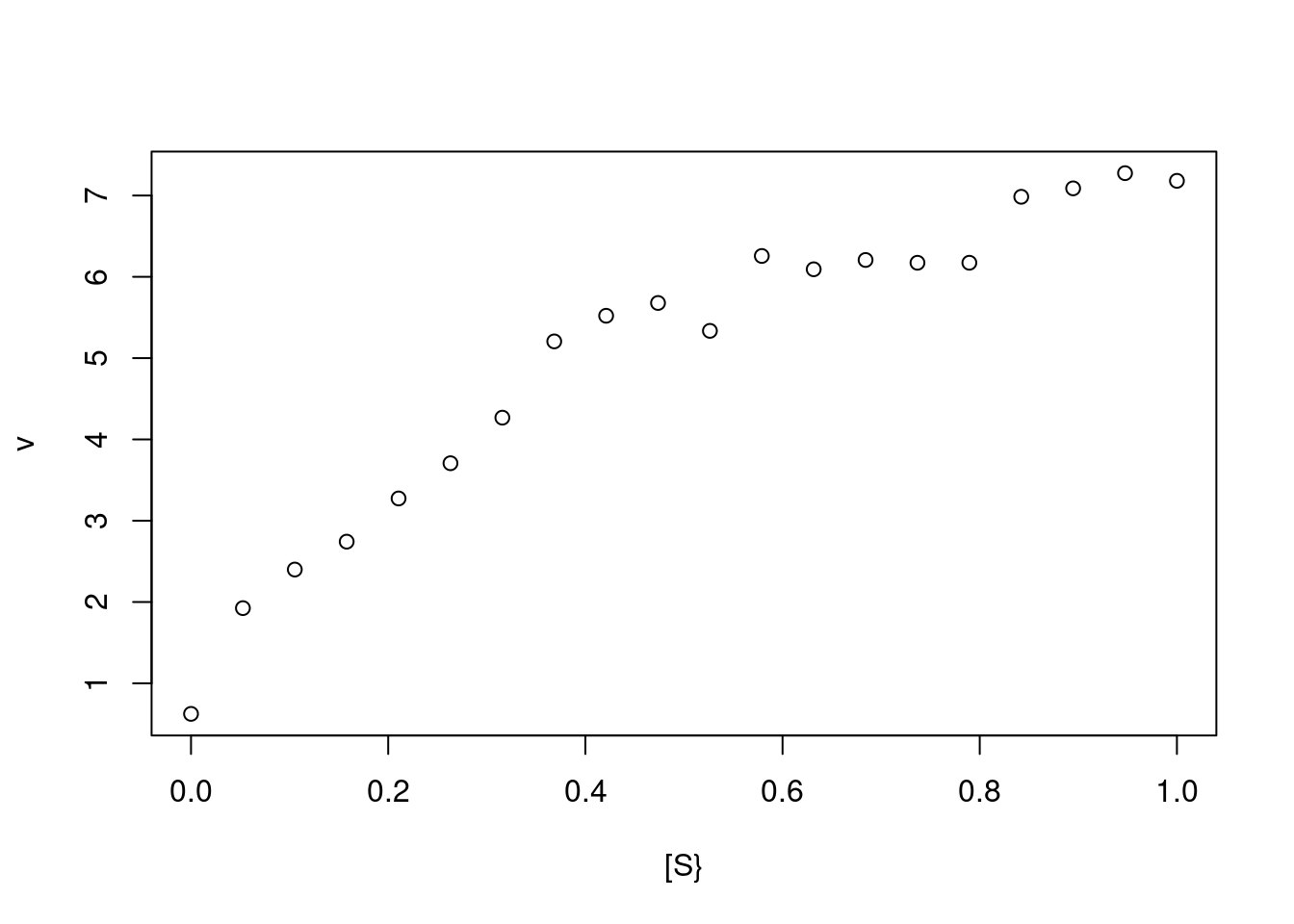
Figura 5.3: Simulação de pontos experimentais (n=20) obtidos a partir da equação de Michaelis-Menten.
5.2 Linearizações e ajustes
R para, respectivamente, estabelecer a área gráfica e sua subdivisão para plotagem em 4 paineis (par e mfrow ou mfcol):S=c(0.1,0.2,0.5,1,5,10,20) # cria um vetor para substrato
Km=0.5;Vm=10 # estabelece os parâmetros enzimáticos
v=Vm*S/(Km+S) # aplica a equação de MM ao vetor de S
par(mfrow=c(2,2)) # estabelece área de plot pra 4 gráficos
plot(S,v,type="o",main="Michaelis-Mentem")
plot(1/S,1/v,type="o",main="Lineweaver-Burk")
plot(v,v/S,type="o",main="Eadie-Hofstee")
plot(S,S/v,type="o",main="Hanes-Woolf")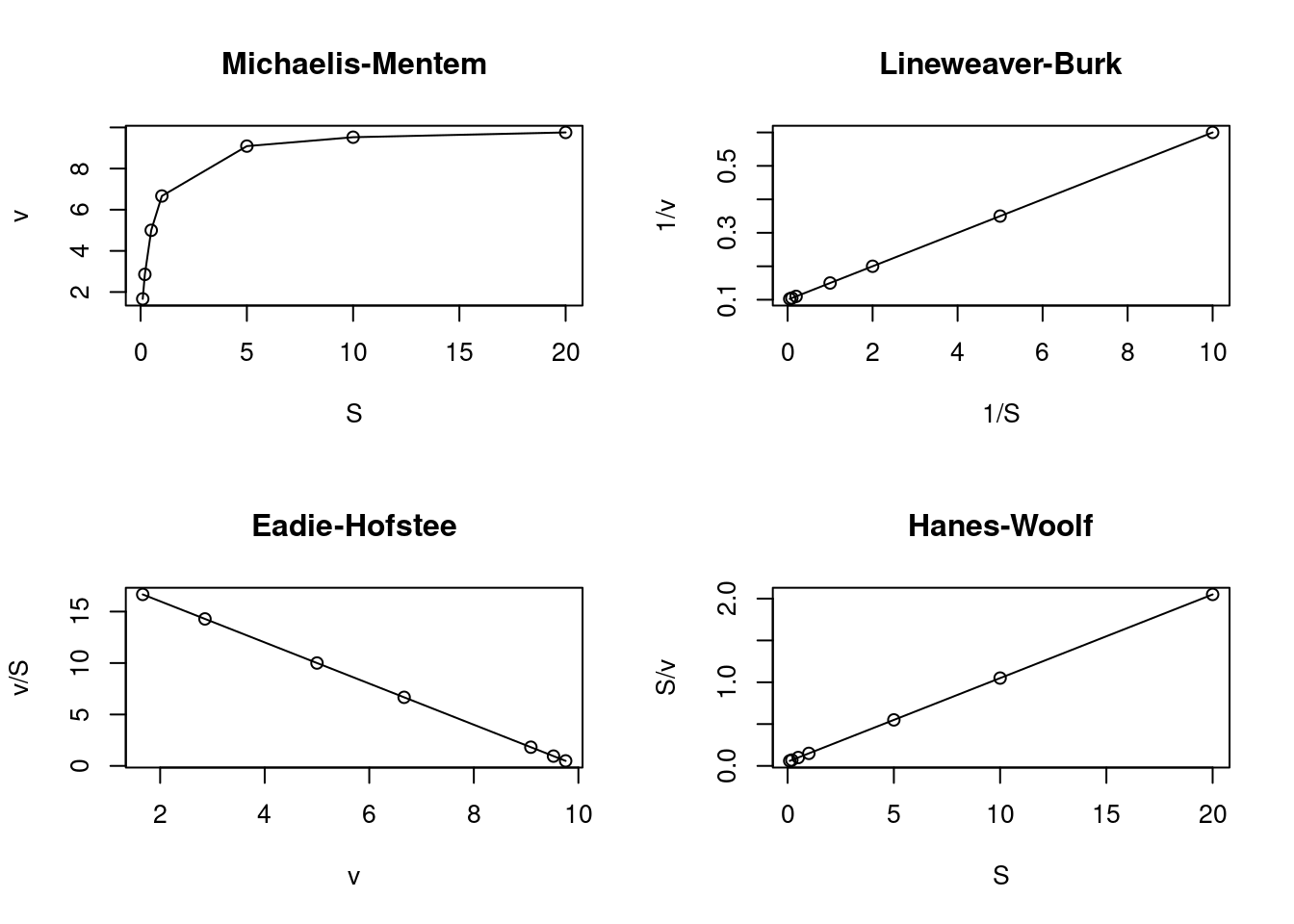
Figura 5.4: Principais linearizações da equação de Michaelis-Menten.
layout(1) # volta à janela gráfica normal5.2.1 Linearização por transformação de Lineweaver-Burk
S=seq(0.1,1,length.out=20) # gera uma sequência com 20 pontos entre 0 e 1 para valores de substrato
Vm=10; Km=0.5 # parâmetros cinéticos
set.seed(1500) # estabelecer a mesma semente aleatória do gráfico direto de Michaelis-Menten, para reproducibilidade dos pontos
erro=runif(20,0,1) # comando para erro uniforme (no. de pontos, min, max)
v=Vm*S/(Km+S)+erro # equação de Michaelis-Menten
inv.S=1/S # cria variáveis para o duplo-recíproco
inv.v=1/v
plot(inv.v~inv.S, xlab="1/S", ylab="1/v") # elabora o gráfico de Lineweaver-Burk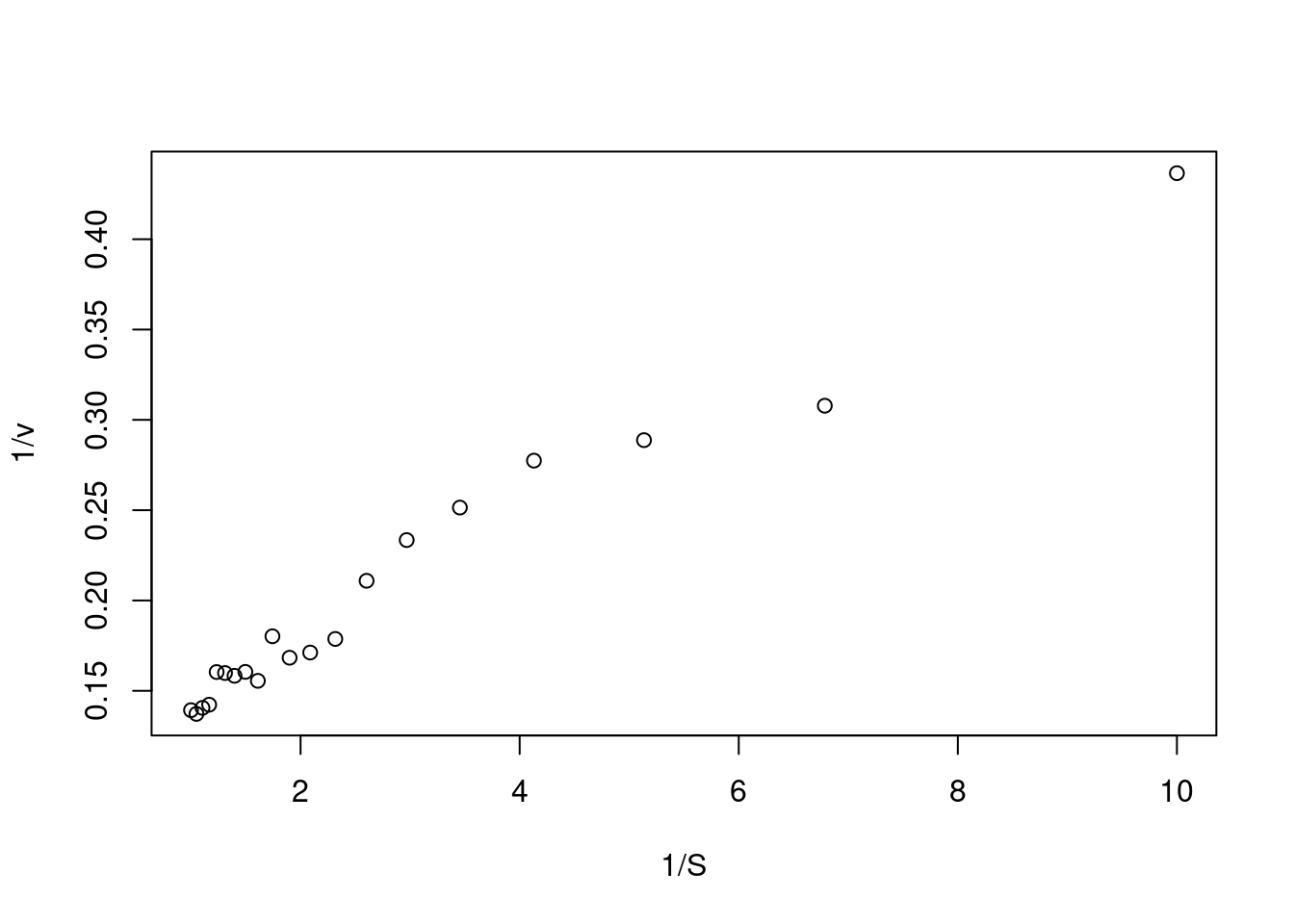
Figura 5.5: Representação de Lineweaver-Burk para os dados simulados da curva de Michaelis-Menten.
R, isso pode ser facilmente conduzido pelo trecho de código (chunk) que segue:reg.LB<-lm(inv.v~inv.S) # expressão para ajuste linear
summary(reg.LB) # resultados do ajuste##
## Call:
## lm(formula = inv.v ~ inv.S)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.02820 -0.00986 -0.00350 0.00748 0.02842
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 0.11363 0.00515 22.1 1.7e-14 ***
## inv.S 0.03277 0.00146 22.4 1.3e-14 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.01 on 18 degrees of freedom
## Multiple R-squared: 0.965, Adjusted R-squared: 0.964
## F-statistic: 503 on 1 and 18 DF, p-value: 1.33e-14plot(inv.v~inv.S, xlab="1/S", ylab="1/v") # gráfico de Lineweaver-Burk
abline(reg.LB, col="blue") # sobreposição do ajuste ao gráfico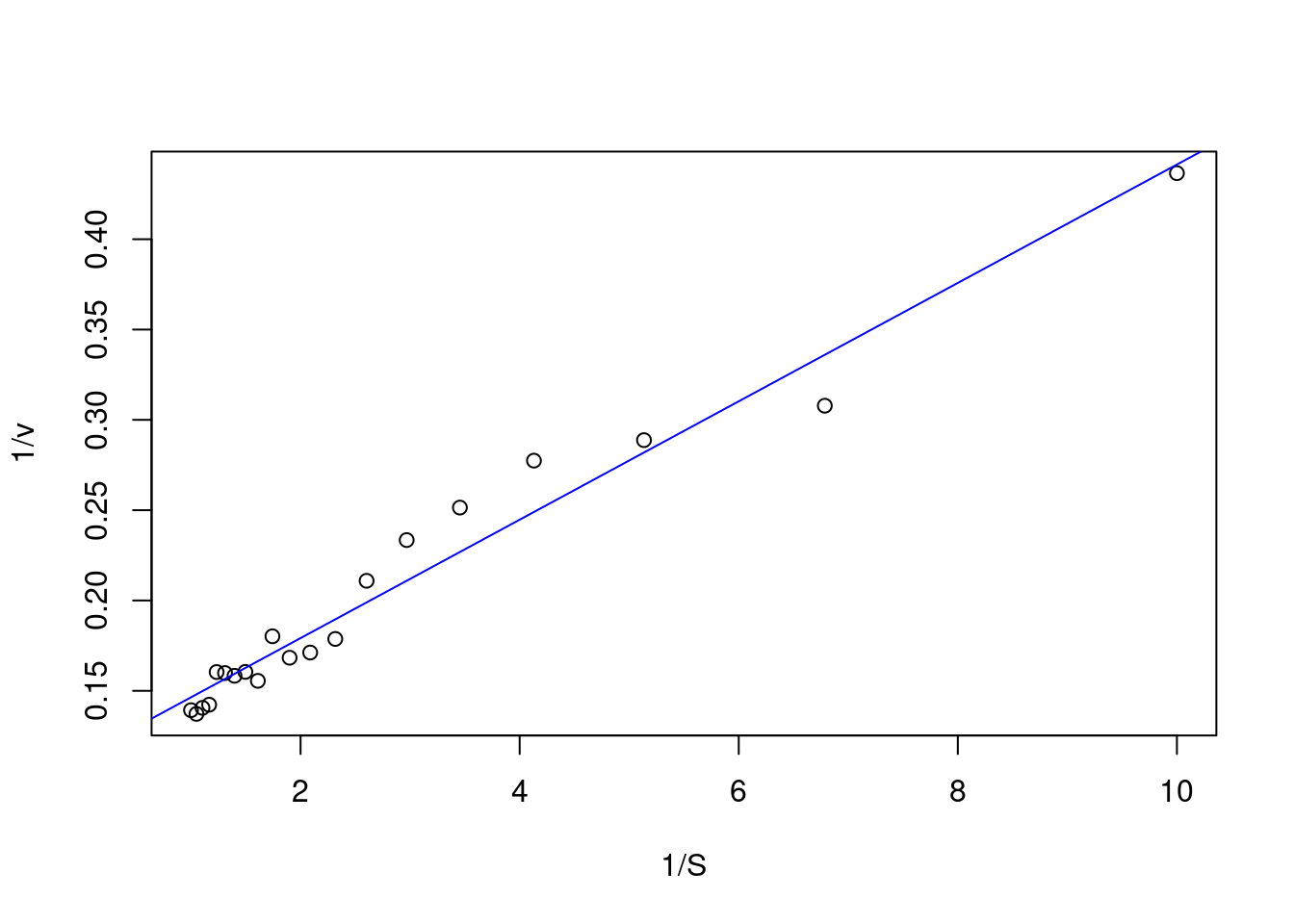
Figura 5.6: Ajuste linear para os dados de Lineweaver-Burk.
R para a função lm de ajuste linear por mínimos quadrados possui diversas informações que nos permite avaliar a qualidade da regressão. Brevemente, esse tabela nos fornece o valor de cada parâmetro do ajuste conforme a equação que segue:valor de erro-padrão dos parâmetros (Std. Error);
valor da distribuição t de Student (t value);
o respectivo nível de probabilidade (Pr) com indicação de significância (asteriscos);
erro padrão residual (Residual standard error);
valor dos coeficientes de determinação bruto (Multiple R-squared) e ajustado para os graus de liberdade (Adjusted R-squared);
valor da distribuição F de Snedocor (F-statistic) de variância do ajuste;
graus de liberdade (DF) e o valor de significância da regressão ao modelo linear obtido pela análise de variância (p-value).
plot(reg.LB) # comando para geração de gráficos diagnósticos de ajuste linear
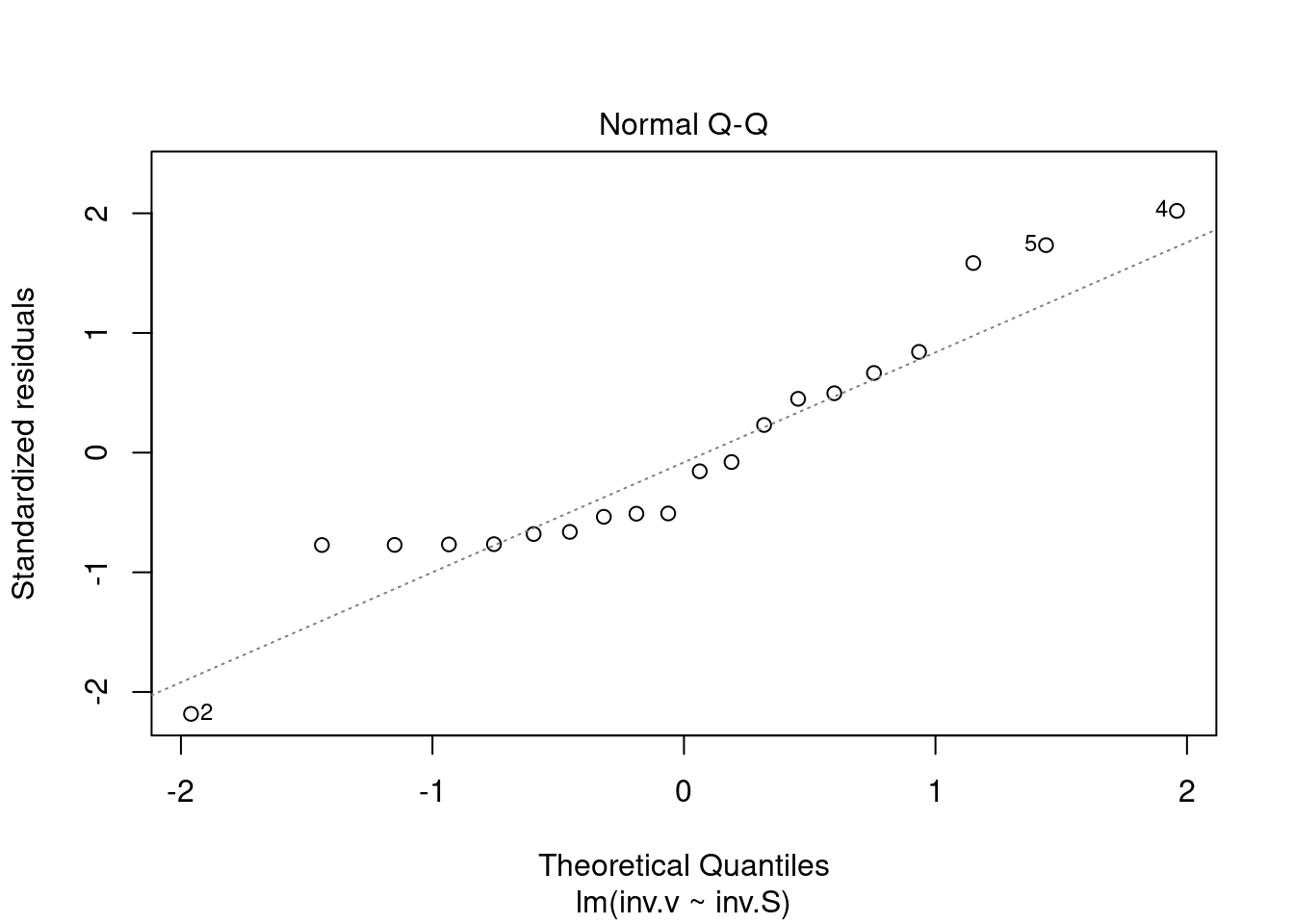
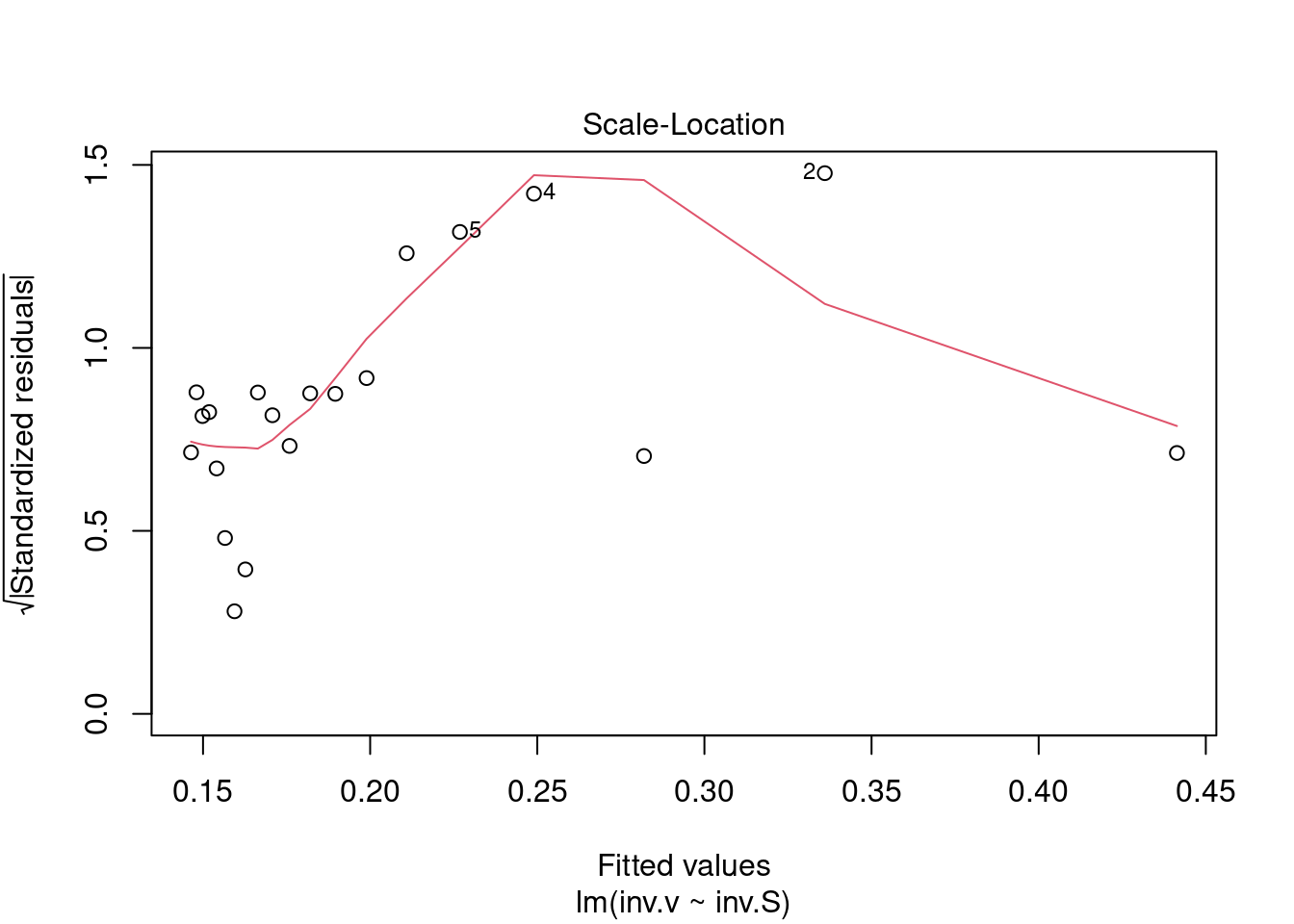
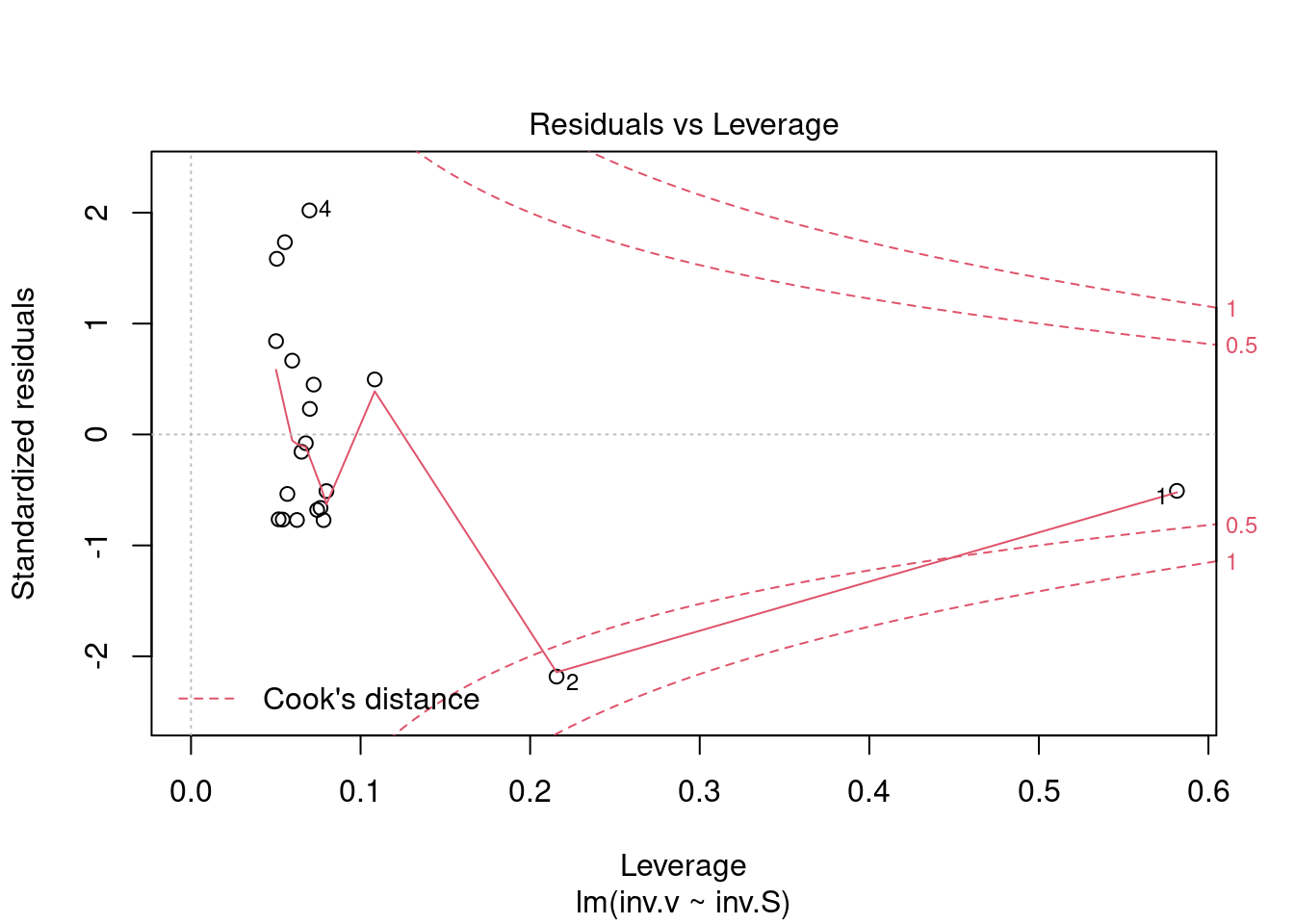
reg.LB<-lm(inv.v~inv.S)
par(mfrow = c(2, 2))
plot(reg.LB)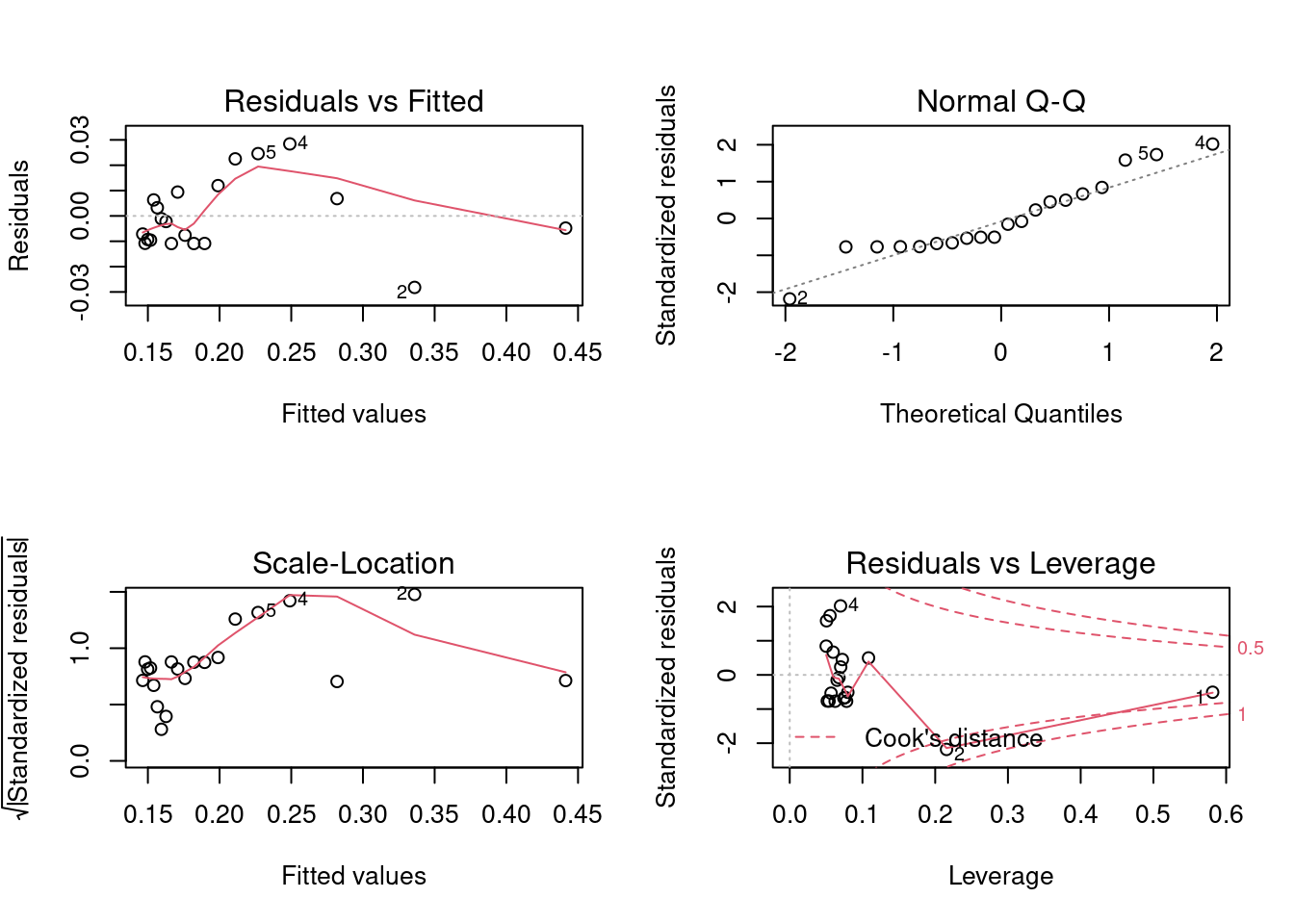
Figura 5.7: Gráficos diagnósticos de ajuste linear.
layout(1)which, 4 ou 6, por ex), tal como em:plot(reg.LB, which = 4)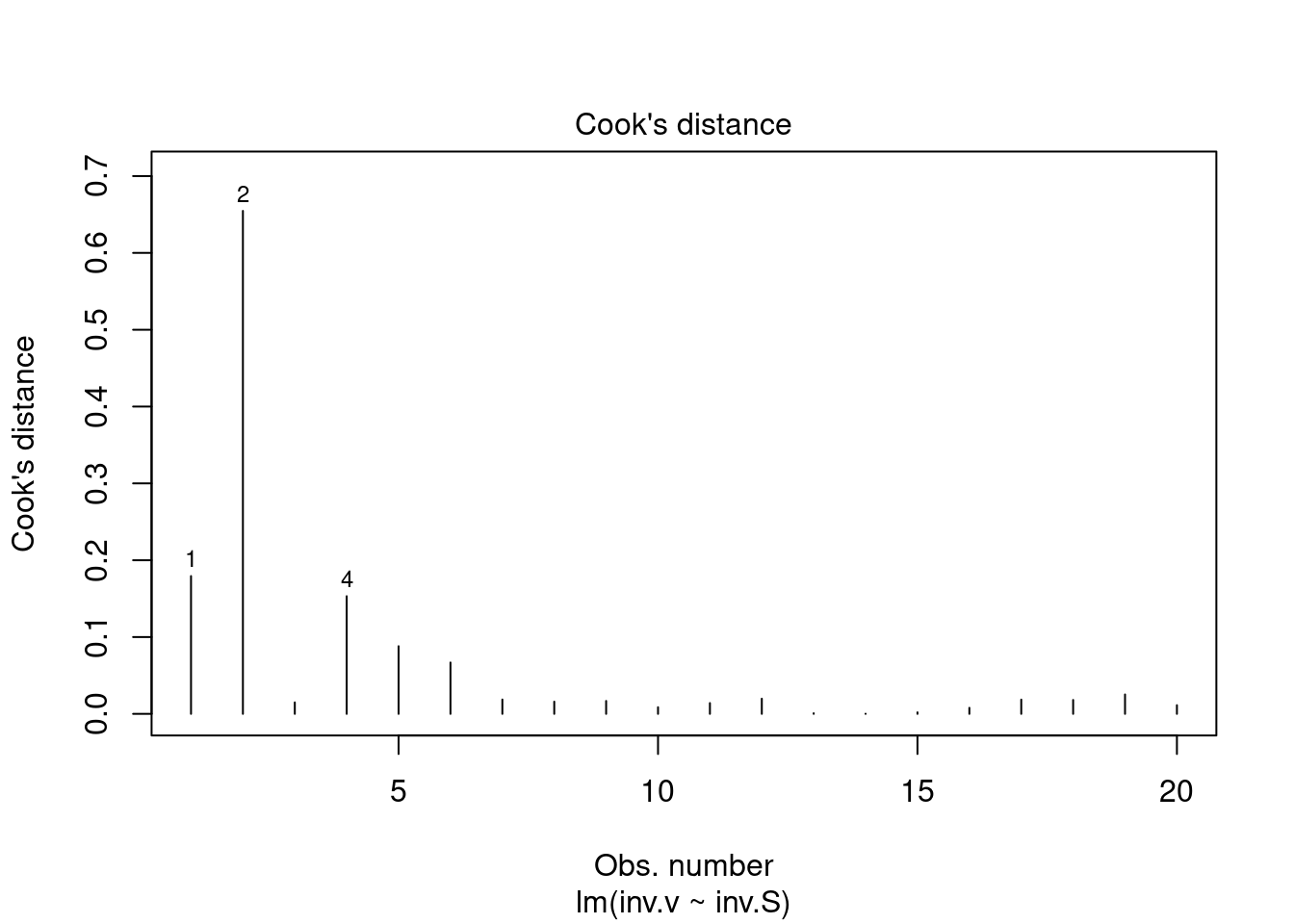
R. Em paralelo, diversas são as funções associadas à própria função lm para modelos lineares (objetos), o que reforça o caráter de linguagem orientada a objeto do R. Entre essas vale citar, com significado intuitivo, coef, fitted, predict, residuals, confint, e deviance.Para acessar os parâmetros contidos na função
lm, assim como outras de mesma natureza no R, basta digitar args:args(lm)## function (formula, data, subset, weights, na.action, method = "qr",
## model = TRUE, x = FALSE, y = FALSE, qr = TRUE, singular.ok = TRUE,
## contrasts = NULL, offset, ...)
## NULLR para diversas situações e tratamentos estatísticos de dados para modelos lineares, e que fogem ao escopo deste manuscrito, tais como os que possibilitam análises de outliers (valores extremos), Generalized Linear Models, Mixed Effects Models, Non-parametric Regression, entre outros. Entre os pacotes do R complementares para regressão linear vale mencionar car, MASS, caret, glmnet, sgd, BLR, e Lars.5.2.1.1 Considerações sobre a linearização por Lineweaver-Burk.
Vm=10; Km=0.5
set.seed(1500) # semente fixa para erro aleatório
erro=runif(length(S),0,0.1)
S=seq(1,10,0.1)
v=Vm*S/(Km+S)+erro## Warning in Vm * S/(Km + S) + erro: longer object length is not a multiple of
## shorter object lengthplot(v~S, xlab="S", ylab="v")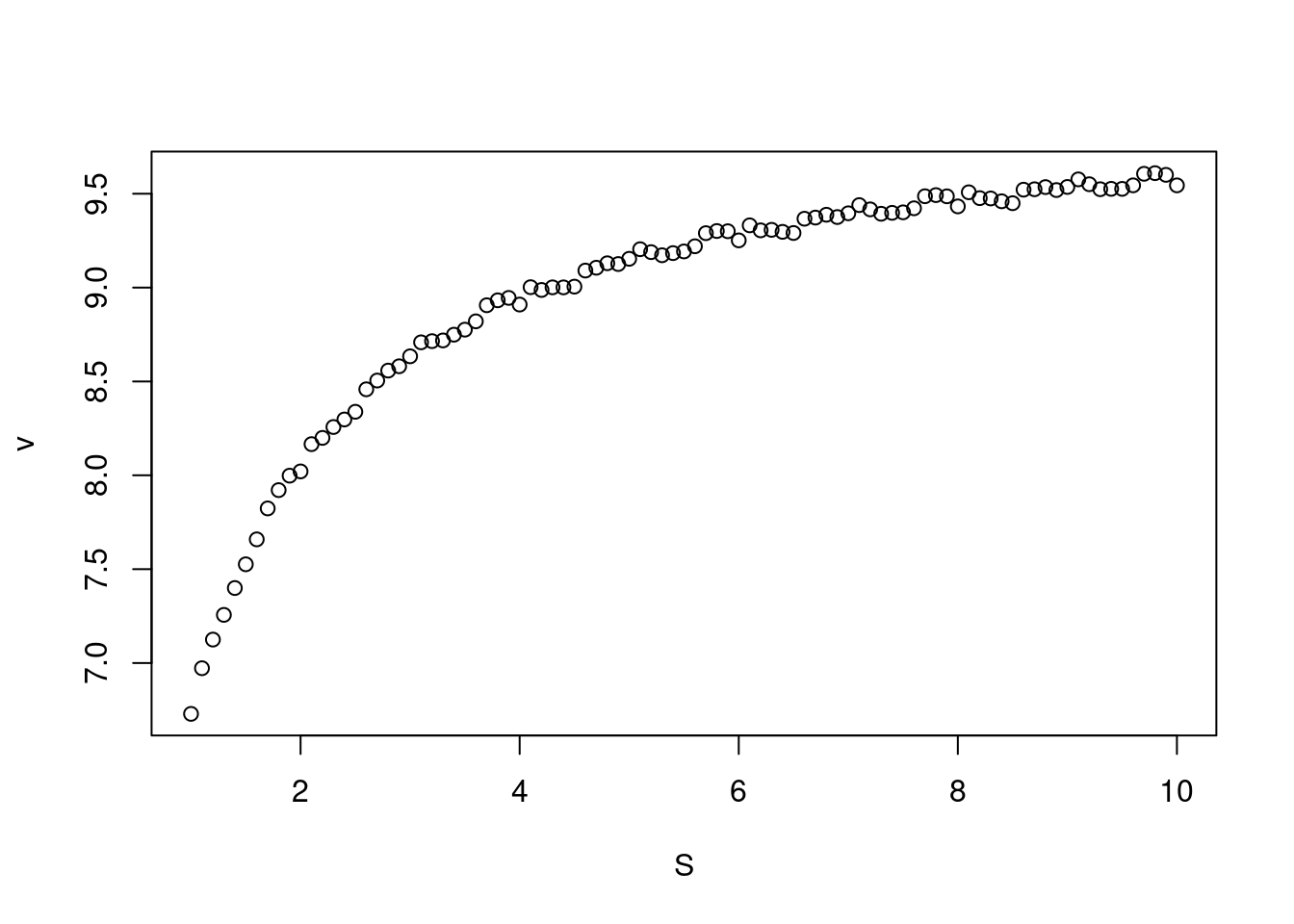
Figura 5.8: Curva de Michaelis-Mentem simulada com erro experimental aleatório. Vm=10; Km=0.5.
set.seed(1500) # semente fixa para erro aleatório
erro=runif(length(S),0,0.2)
Vm=10; Km=0.5 # parâmetros cinéticos
inv.S=1/seq(1,10,0.1) # 1/S
inv.v=1/(Vm*S/(Km+S)+erro) # 1/v
plot(inv.S,inv.v)
lm.LB2<-lm(inv.v~inv.S) # ajuste linear
summary(lm.LB2) #resultados do ajuste##
## Call:
## lm(formula = inv.v ~ inv.S)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.001505 -0.000561 -0.000146 0.000752 0.001412
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 0.099181 0.000136 731 <2e-16 ***
## inv.S 0.048156 0.000419 115 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 8e-04 on 89 degrees of freedom
## Multiple R-squared: 0.993, Adjusted R-squared: 0.993
## F-statistic: 1.32e+04 on 1 and 89 DF, p-value: <2e-16abline(lm.LB2, col="blue")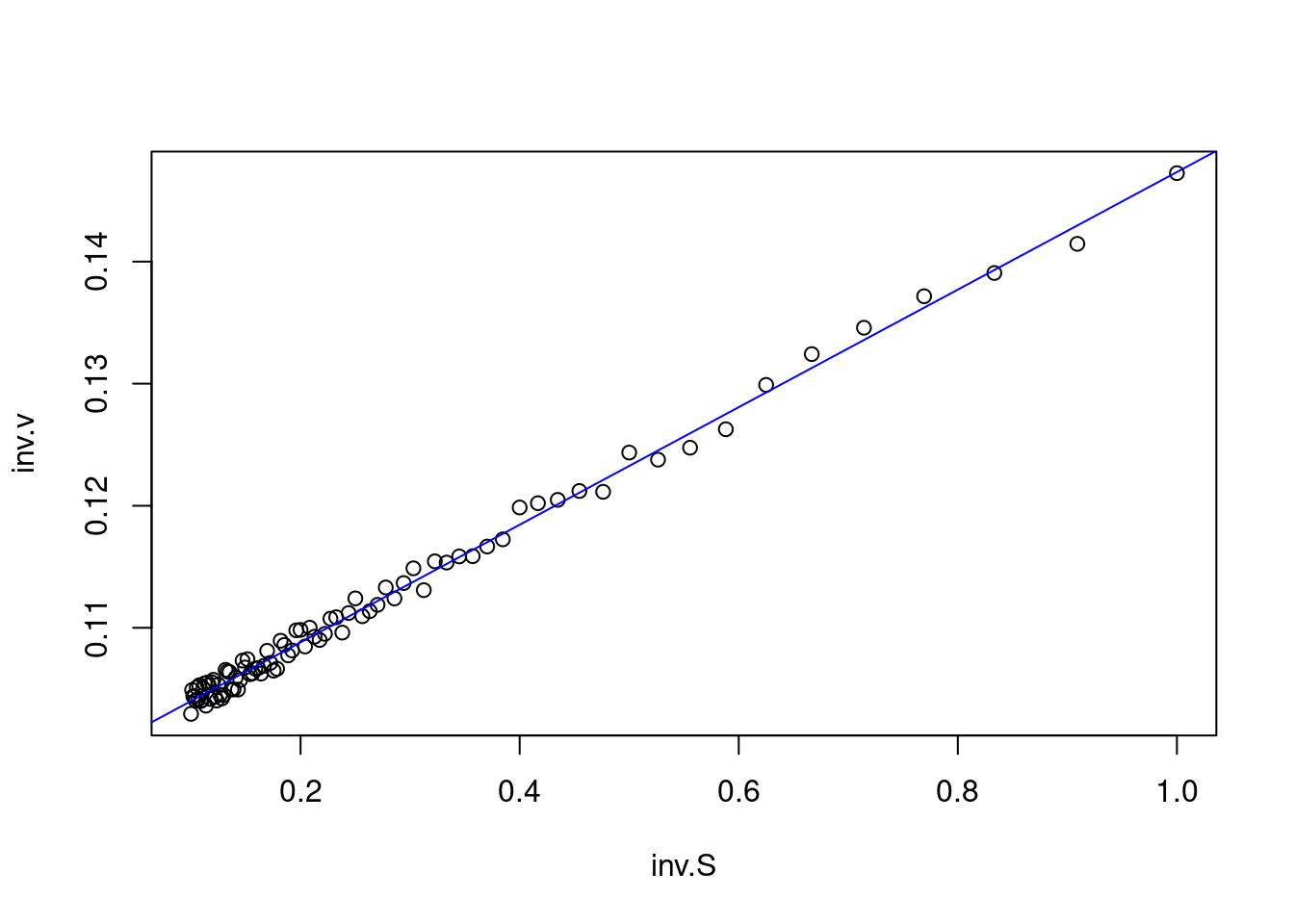
Figura 5.9: Lineweaver-Burk dos dados do gráfico anterior.
Isso pode ser ilustrado variando-se a faixa de valores de S iterativamente, e inspecionando-se o gráfico duplo-recíproco resultante como no trecho de código que segue, e para os mesmos valores da figura 5.9.
set.seed(1500) # mesma semente aleatória para reproducibilidade de erro
Vm=10; Km=0.5 # estabelece os parâmetros de MM
S=seq(10,100,10) # cria-se uma sequência inicial para S
v=Vm*S/(Km+S) # aplicação equação de MM à S
plot(1/S,1/v, type="n",ylim=c(0.098,0.106)) # elabora o duplo-recíproco sem pontos
for(i in 1:3) { # inicia a iteração para gráficos de Lineweaver-Burk
S=seq(10*i,100,length.out=100) # gera uma sequência S com 100 pontos, produzindo 5 vetores que iniciam em valores diferentes para S (10, 30 e 50)
erro=runif(length(S),0,0.1) # erro para adição à vetor de velocidade inicial, com no. de pontos em função do vetor de S
add<-if(i == 1) FALSE else TRUE # controle de fluxo para plotagem de pontos no gráfico vazio
inv.S=1/S; inv.v=1/((Vm*S/(Km+S))+erro) # novos valores para o duplo-recíproco em função da iteração
points(inv.v~inv.S, xlab="1/S", ylab="1/v", col=i, add=add) # adição de pontos ao gráfico de Lineweaver-Burk, com identificação por cores (1, 2, 3, 4 e 5)
lm.LB<-lm(inv.v~inv.S) # elabora o ajuste linear
abline(lm.LB, col=i, lty=i) # sobrepõe as linhas de ajuste
}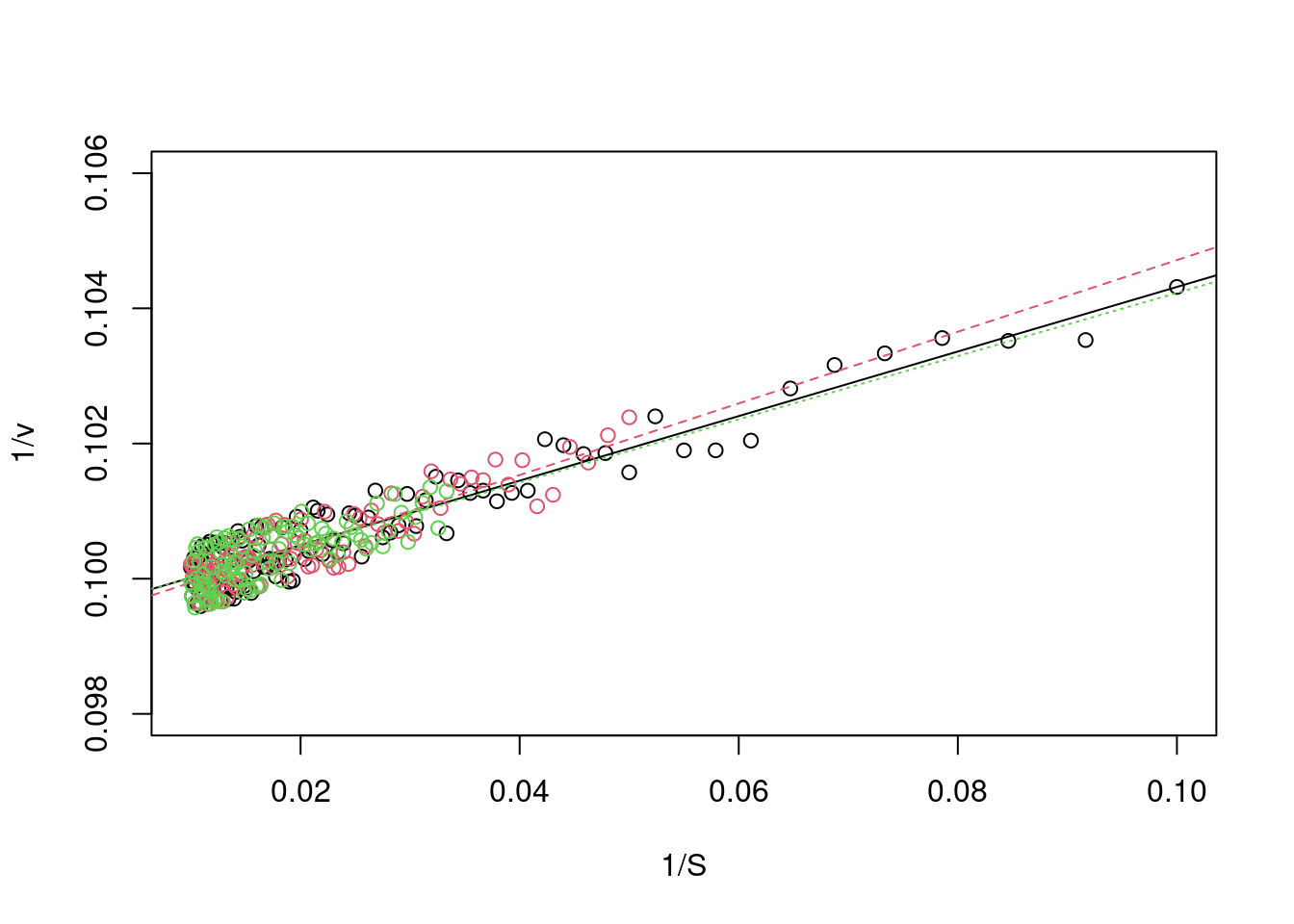
Figura 5.10: Gráficos duplo-recíproco para a curva simulada de Michaelis-Menten, para distintos teores de S inicial.
5.2.2 Linearização por transformação de Eadie-Hofstee
Vm=10; Km=0.5
set.seed(1500) # semente fixa para erro aleatório
erro=runif(length(S),0,0.1)
S=seq(1,10,0.1)
v=Vm*S/(Km+S)+erro
v.S=v/S
plot(v.S~v, xlab="v", ylab="v/S")
lm_EH<-lm(v.S~v); summary(lm_EH)##
## Call:
## lm(formula = v.S ~ v)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.1160 -0.0650 0.0117 0.0546 0.1248
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 20.31073 0.07906 257 <2e-16 ***
## v -2.02191 0.00893 -226 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.07 on 98 degrees of freedom
## Multiple R-squared: 0.998, Adjusted R-squared: 0.998
## F-statistic: 5.13e+04 on 1 and 98 DF, p-value: <2e-16abline(lm_EH, col="blue")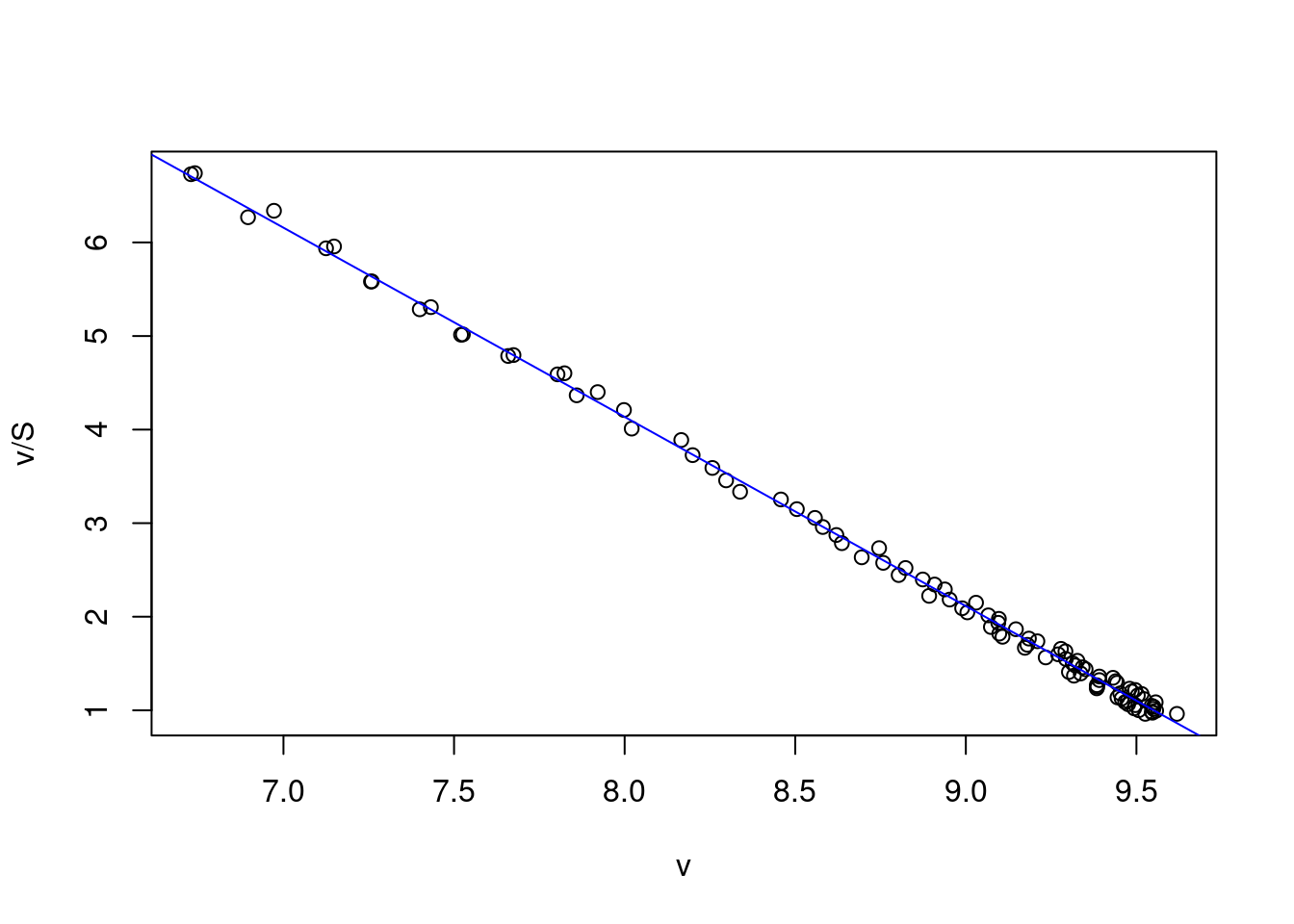
5.2.3 Distribuição de erros nas linearizações de Michaelis-Mentem
O código abaixo ilustra a distribuição de erros dessas transformações, introduzindo uma função importante do
R para construção de gráficos com barras de erros: arrows.Vm=10; Km=0.5 # fixa os parâmetros de MM
set.seed(1500) # fixa semente para erro aleatório
erro=runif(length(S),0,0.5) # vetor de erro uniforme
S=c(0.1,0.2,0.5,1,5,10,20) # vetor de substrato
v=Vm*S/(Km+S) # equação de MM
par(mfrow=c(2,2)) # área de plot pra 4 gráficos
plot(S,v,type="o",main="Michaelis-Mentem")
arrows(S,v,S,v-erro,length=.05,angle=90) # barra inferior de erro
arrows(S,v,S,v+erro,length=.05,angle=90) # barra superior de erro
plot(1/S,1/v,type="o",main="Lineweaver-Burk")
arrows(1/S,1/v,1/S,1/(v-erro),length=.05,angle=90)
arrows(1/S,1/v,1/S,1/(v+erro),length=.05,angle=90)
plot(v,v/S,type="o",main="Eadie-Hofstee")
arrows(v,v/S,v,(v-erro)/S,length=.05,angle=90)
arrows(v,v/S,v,(v+erro)/S,length=.05,angle=90)
plot(S,S/v,type="o",main="Hanes-Woolf")
arrows(S,S/v,S,S/(v-erro),length=.05,angle=90)
arrows(S,S/v,S,S/(v+erro),length=.05,angle=90)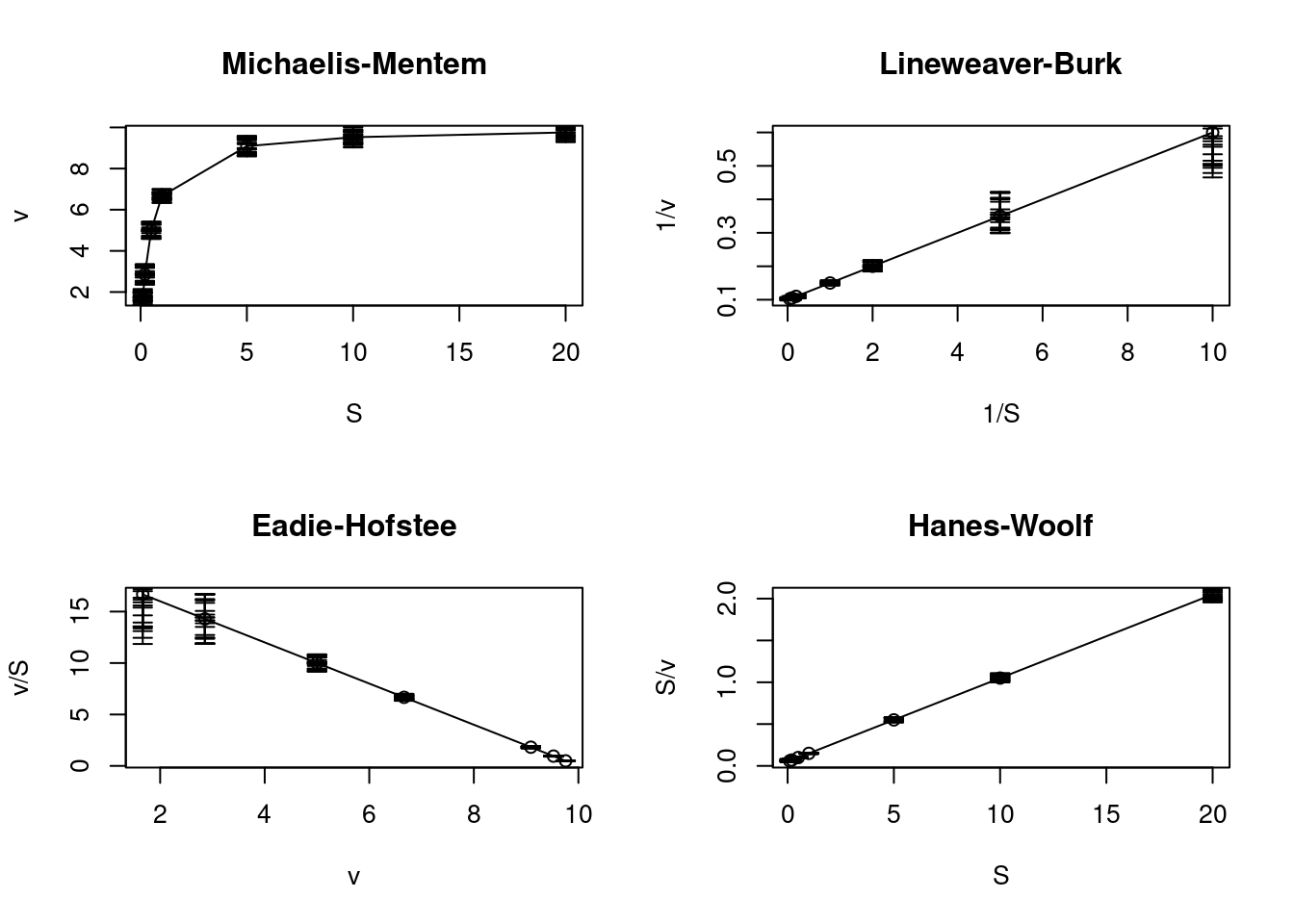
Figura 5.11: Distribuição de erros aleatórios para a equação de Michaelis-Mentem e suas transformações lineares.
par(mfrow=c(1,1)) # retorno à janela gráfica normalAinda que a representação dos duplos-recíprocos tenha em si algumas desvantagens, tais como a dispersão de erros principalmente em valores baixos de S, é a única dentre as mencionadas que permite um ajuste linear por mínimos quadrados, se considerarmos as premissas estatísticas desse.
Para que se possa obter parâmetros de intercepto e inclinação a partir de uma regressão linear, é necessário que se cumpra as premissas estatísticas de 1) distribuição normal de resíduos, 2) homogeneidade de variâncias, e 3) independência das variáveis. Se observarmos as três linearizações, tanto a de Eadie-Hofstee como a de Hanes-Woolf não cumprem a premissa de independência, já que a variável dependente (y) é função da independente (x).
Para que uma transformação por duplos-recíprocos possa ser utilizada mais fielmente à obtenção de parâmetros cinéticos, contudo, pode-se adotar o cômputo de peso na fórmula de ajuste linear, tal como sugerido por Wilkinson (Wilkinson 1961), considerando-o como o recíproco das variâncias estimadas. Nesse caso, o ajuste linear considerando o quadrado do vetor de erros aleatórios como variância e o peso como seu recíproco (1/s²), pode ser esboçado como:
S=seq(0.1,1,length.out=20)
Vm=10; Km=0.5
set.seed(1500)
erro=runif(20,0,1)
v=Vm*S/(Km+S)+erro
inv.S=1/S
inv.v=1/v
reg.LB.peso<-lm(inv.v~inv.S, weights=1/erro^2) # expressão para ajuste linear
summary(reg.LB.peso) # resultados do ajuste##
## Call:
## lm(formula = inv.v ~ inv.S, weights = 1/erro^2)
##
## Weighted Residuals:
## Min 1Q Median 3Q Max
## -0.04779 -0.02231 -0.01849 0.00162 0.04830
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 0.11733 0.00254 46.1 < 2e-16 ***
## inv.S 0.03491 0.00145 24.0 3.9e-15 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.03 on 18 degrees of freedom
## Multiple R-squared: 0.97, Adjusted R-squared: 0.968
## F-statistic: 578 on 1 and 18 DF, p-value: 3.93e-15Complementarmente, pode-se obter uma comparação estatística entre o modelo linear simples e o que adotou o peso estatístico por:
anova(reg.LB,reg.LB.peso)## Analysis of Variance Table
##
## Model 1: inv.v ~ inv.S
## Model 2: inv.v ~ inv.S
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 18 0.00383
## 2 18 0.01585 0 -0.0125.3 Ajuste não-linear
O ajuste não-linear difere do linear em algumas características, tais como:
- A busca iterativa de um valor mínimo (local ou global) para a soma dos quadrados dos erros das estimativas;
- a necessidade de um valor inicial para os parâmetros (sementes);
- a linearidade nos erros e no gradiente da função sobre os parâmetros.
- a necessidade de algoritmo mais sofisticado para solução simbólica e matricial para minimizar a derivada da função sobre cada parâmetro;
- a necessidade de programa que trabalhe com álgebra matricial (computador, dispositivo móvel ou calculadora);
- o uso de algoritmos mais sofisticados (Gauss, Newton-Raphson, Levenberg-Marquadt, Simplex).
- o emprego da equação original do modelo, por vezes de difícil linearização.
5.3.1 Ajuste não-linear da equação de Michaelis-Mentem
Vm=10
Km=0.5
set.seed(1500)
erro=runif(20,0,1)
S=seq(0,1,length.out=20)
v=Vm*S/(Km+S)+erro
dat.Sv <- data.frame(S,v) # criação de planilha com S e v
plot(v~S, type="p", from = 0, to=1, n = 20,
xlab="[S}", ylab="v") # construção do gráfico de MM
nl.MM<-nls(v~Vm*S/(Km+S), start = list(Vm=7, Km=0.2), data=dat.Sv) # linha de código para ajuste não linear
lines(S,fitted(nl.MM),col="red") # sobreposição da linha ajustada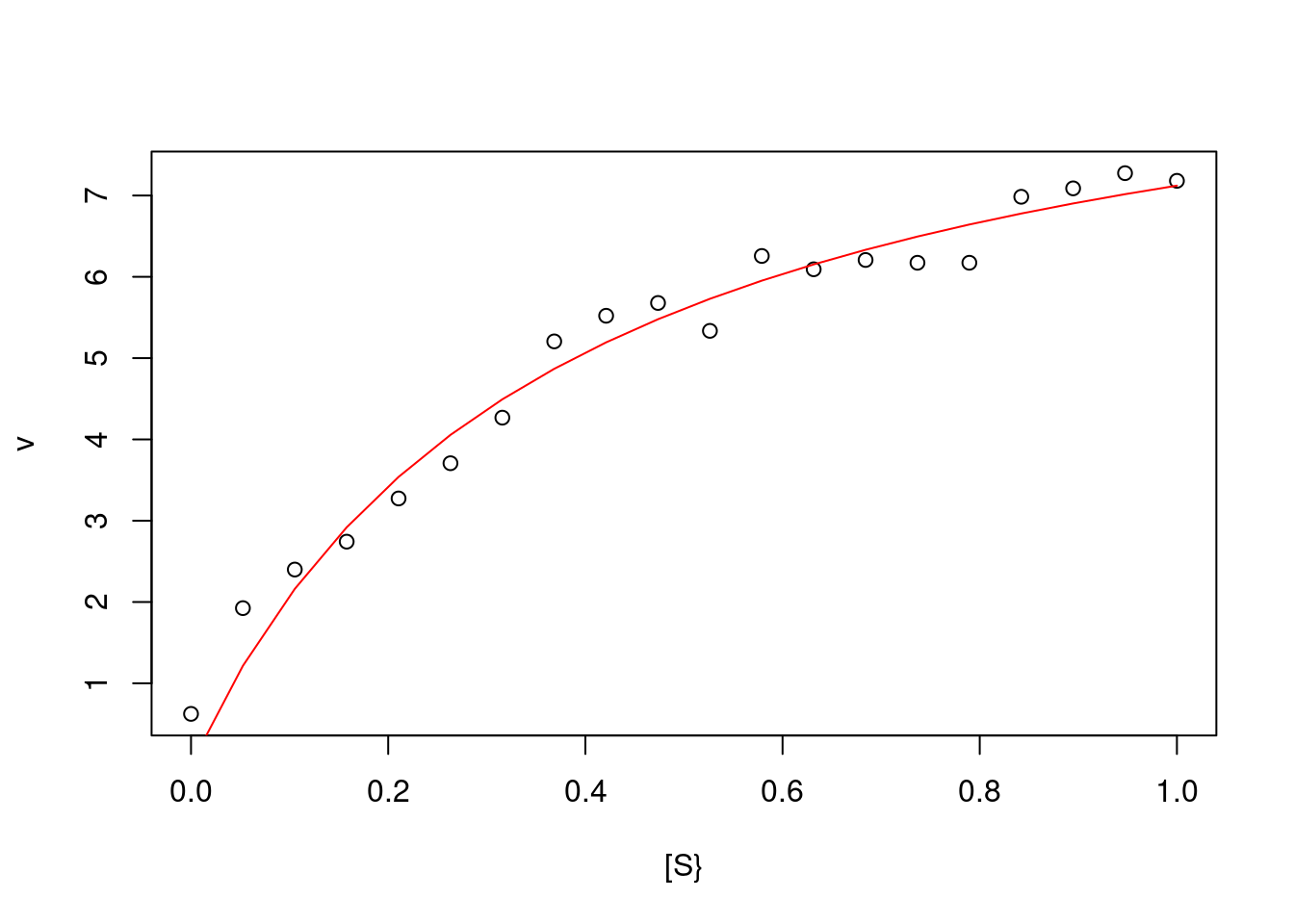
Figura 5.12: Ajuste não linear para a simulação experimental da equação de Michaelis-Menten.
summary(nl.MM) # sumário dos resultados##
## Formula: v ~ Vm * S/(Km + S)
##
## Parameters:
## Estimate Std. Error t value Pr(>|t|)
## Vm 9.7549 0.5211 18.72 3.0e-13 ***
## Km 0.3698 0.0502 7.37 7.7e-07 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.4 on 18 degrees of freedom
##
## Number of iterations to convergence: 5
## Achieved convergence tolerance: 1.63e-06Assim como para ajuste linear, os parâmetros que abrangem a função
nls envolvem:args(nls)## function (formula, data = parent.frame(), start, control = nls.control(),
## algorithm = c("default", "plinear", "port"), trace = FALSE,
## subset, weights, na.action, model = FALSE, lower = -Inf,
## upper = Inf, ...)
## NULLR e que permite ajustes não-lineares (stats), existem diversos outros que permitem ajustes com algoritmos, avaliações e plotagens variadas, tais como nlme (mixed-effects), nlrwr, nlstools, nls2, nls.multstart, minpack.lm (algoritmo de Levenberg-Marquadt), nlshelper, e nlsLM.5.3.2 Algumas vantagens do modelo linear sobre o não-linear
- É mais fácil, com algoritmo simplificado, e mesmo pelo uso de somatórias de algumas quantidades envolvendo x e y, sendo resolvido com calculadora científica simples, ou mesmo à mão;
- é mais intuitivo visualmente, posto que o modelo final será sempre uma reta;
- possui apenas dois parâmetros na equação, intercepto e inclinação;
- requer poucas medidas, já que uma reta se constroi com apenas dois pontos;
- não requer sementes para estimativas iniciais o que, a depender do modelo não-linear, pode ser bem abstrato, culminando em mínimos locais ou mesmo na falta de solução para o ajuste;
- permite interpretação experimental quando há fuga da linearidade;
- independe de um modelo físico específico;
- não requer, por vezes, a necessidade de constrição de resultados (constraints), por exemplo instruindo o algoritmo a buscar uma estimativa obrigatoriamente de valor positivo para o parâmetro.
- relações lineares e transformações são encontradas em inúmeros modelos físicos nas Ciências Naturais.
5.4 Enzimas alostéricas
A equação que define uma enzima alostérica em função do teor de seu substrato dada abaixo:
Onde nH representa o coeficiente de cooperatividade ou constante de Hill para a ligação com moléculas de S (de maneira similar à ligação de \(O_{2}\) à hemoglobina. De modo geral, o valor de nH pode ser inferior à unidade (cooperatividade negativa) ou superior a essa (cooperatividade positiva). Para ilustrar o comportamento cinético de uma enzima alostérica, segue o trecho abaixo, que também introduz outro formato para representar curvas no R nomeando a variável independente (x).
v = function(S, Vm=10, Km=3, nH=2) {Vm*S^nH/(Km^nH+S^nH)}
curve(v,from=0,to=10,n=100, xlab="S", ylab="v",
bty="L") # eixos em L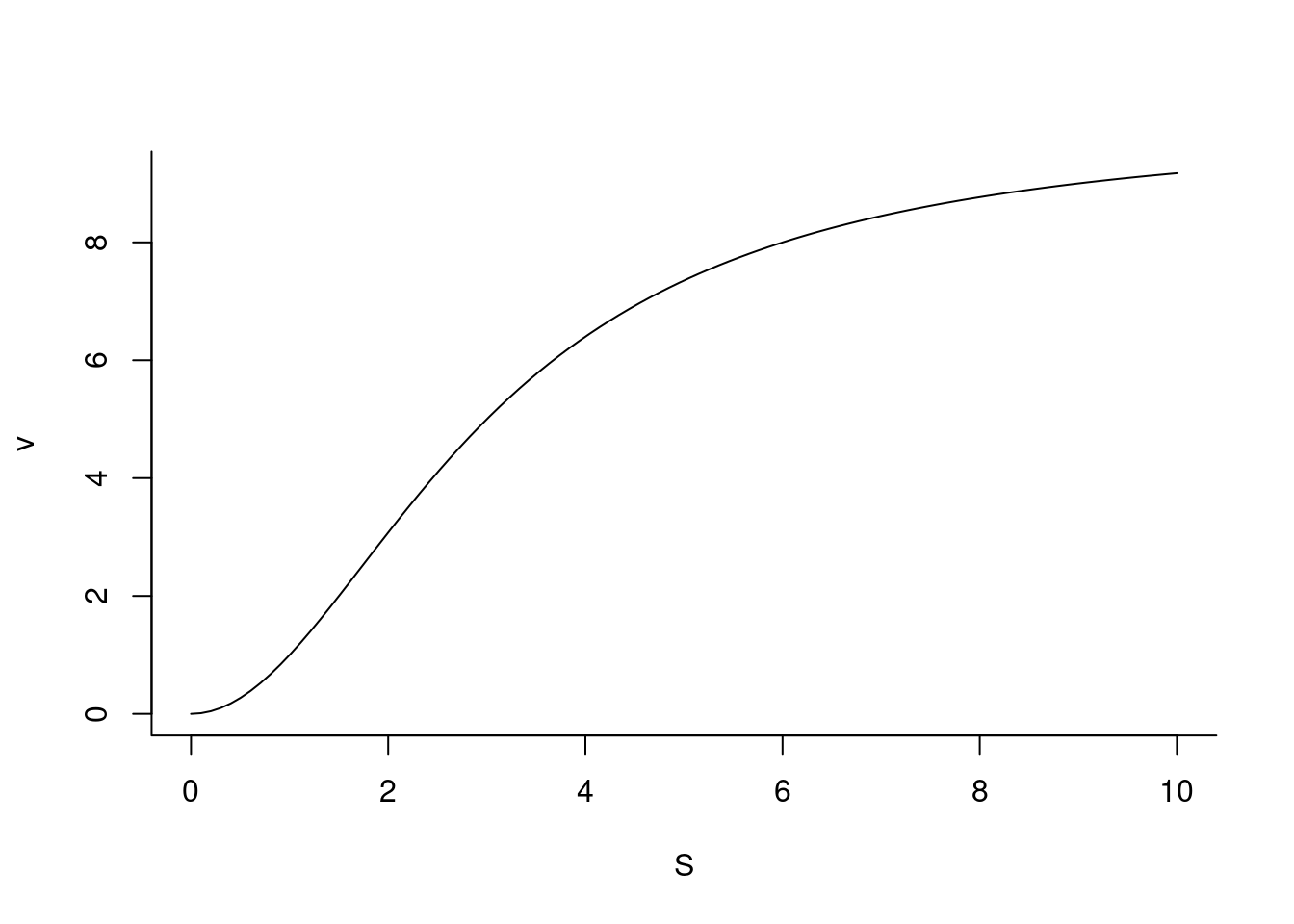
nH=seq(from=0.1,to=3,length.out=7) # sequência para 7 valores de nH
for(i in 1:length(nH)) { # loop para adicionar curva alostérica a cada valor de nH
add<-if(i == 1) FALSE else TRUE # controle de fluxo
v = function(S, Vm=10, Km=3, a=nH[i]) {Vm*S^a/(Km^a+S^a)}
curve(v,from=0,to=4, n=500, col=i, xlab="S", ylab="v",
bty="L", add=add)
}
arrows(0,5,3,2,length=0.1,angle=45, col="blue") # seta para nH
text(0.5,5.2,"nH",col="blue") # indexador para nH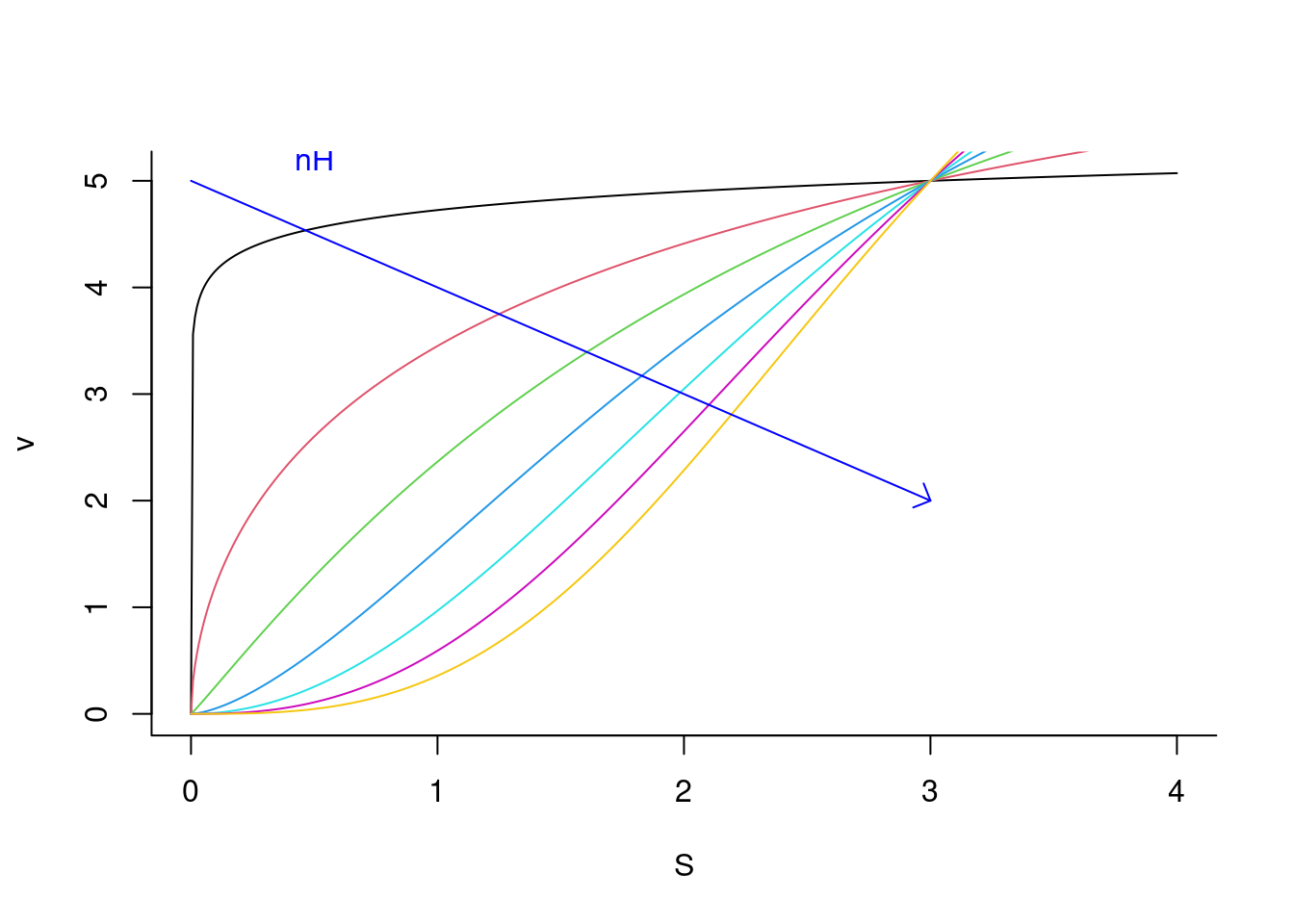
Figura 5.13: Perfil alostérico de uma enzima sob variação do coeficiente de cooperatividade nH.
5.5 Inibição Enzimática
5.5.1 Inibição pelo substrato
S=seq(0,10,0.1)
v_alos <- function(S, Vm=10, Km=0.5, Ks=2) {Vm*S/(S*(1+S/Ks)+Km)}
curve(v_alos, xlim=c(0,10), xlab="S",ylab="v")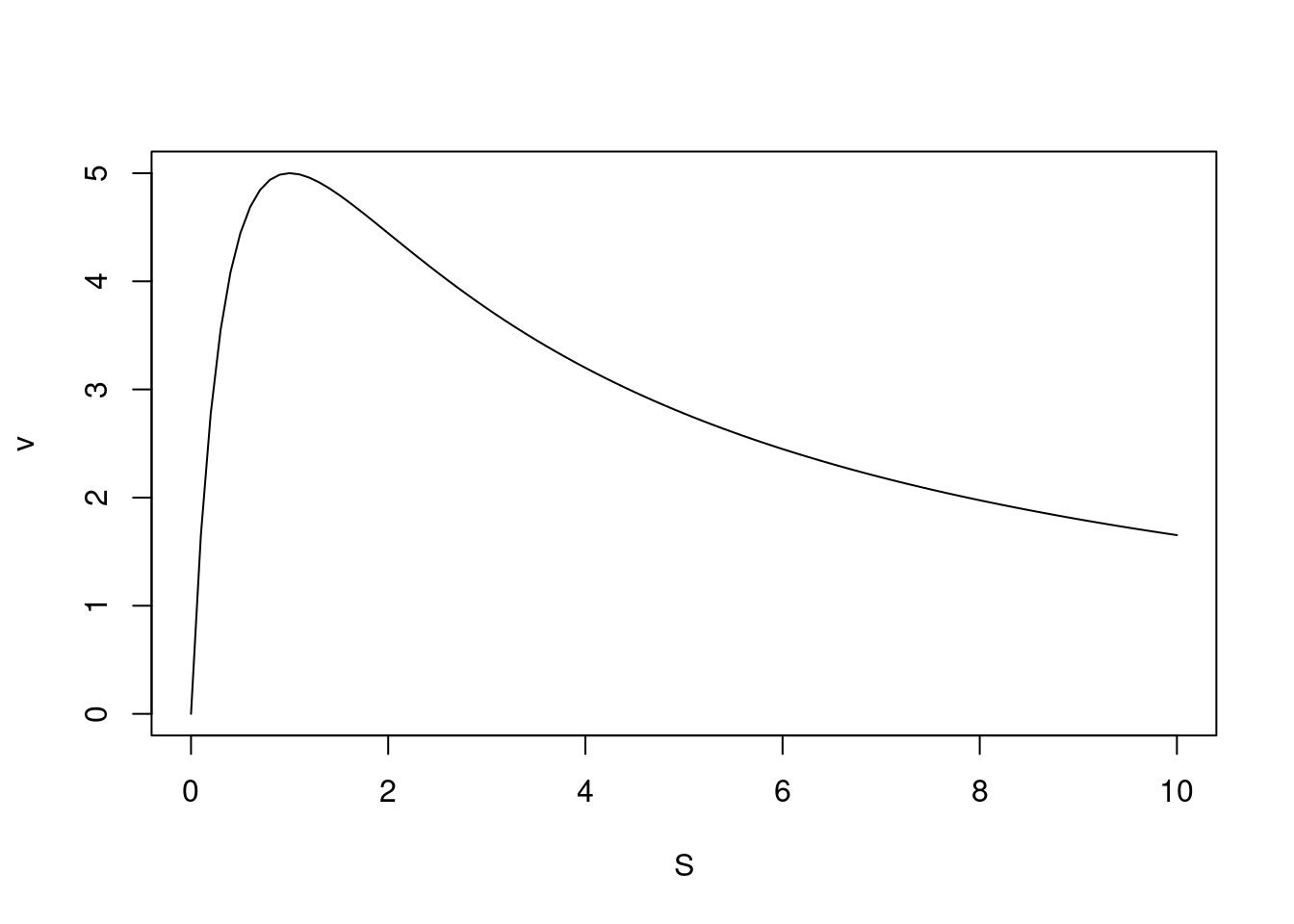
Figura 5.14: Comportamento cinético de uma enzima inibida por excesso de substrato.
5.5.2 Modelos de inibição enzimática
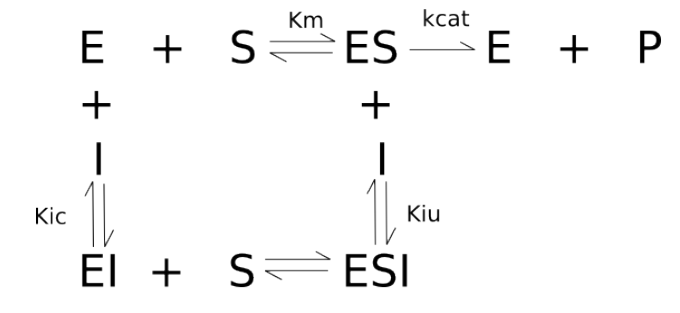
Figura 5.15: Diagrama representativo dos tipos de inibição enzimática. kcat: constante catalítica; Ki: constante de equilíbrio de dissociação do inibidor, com índices para inibição competitiva (Ki), incompetitiva (Kiu) e não competitiva (Kic e Kiu).
Um modelo matemático que abrange esses três tipos de inibição enzimática é descrito na equação abaixo:
5.5.2.1 Curva de Michaelis-Mentem para modelos de inibição enzimática
R as cuvas michaelianas para modelos clássicos de inibição, considerando valores para as constantes de equilíbrio de dissociação dos inibidores como Kic = 0.2, e Kiu = 1, como no trecho de código abaixo.par(mfrow=c(2,2)) # divide a área de plotagem
S=seq(0,10,0.1) # geração de teores de S
contr <- function(S,Vm=10,Km=0.5) {Vm*S/(Km+S)} # função de MM, sem inibição
curve(contr, xlim=c(0,10), xlab="S",ylab="v",main="Competitiva") # cuva controle; veja que o título tem que ser adicionado para a 1a. de par de curvas, controle e inibição
# Modelos de inibição:
# Competitiva
comp.i <- function(S, Vm=10, Km=0.5, I=2, Kic=0.2) {Vm*S/(Km*(1+I/Kic)+S)}
curve(comp.i, add = TRUE, col="red",lty=2) # competitiva
# Não competitiva pura
pura.i <- function(S, Vm=10, Km=0.5, I=2, Ki=1) {Vm*S/(Km*(1+I/Ki)+S*(1+I/Ki))}
curve(contr, xlim=c(0,10), xlab="S",ylab="v",main="Não Compet. Pura")
curve(pura.i, add = TRUE, col="red",lty=2) # não competitiva pura (Kiu=Kic=Ki)
# Não competitiva mista
mista.i <- function(S, Vm=10, Km=0.5, I=2, Kic=0.2, Kiu=1) {Vm*S/(Km*(1+I/Kic)+S*(1+I/Kiu))}
curve(contr, xlim=c(0,10), xlab="S",ylab="v",main="Não Compet. Mista")
curve(mista.i, add = TRUE, col="red",lty=2) # não competitiva mista
# Incompetitiva
incomp.i <- function(S, Vm=10, Km=0.5, I=2, Kiu=1) {Vm*S/(Km+S*(1+I/Kiu))}
curve(contr, xlim=c(0,10), xlab="S",ylab="v",main="Incompetitiva")
curve(incomp.i, add = TRUE, col="red",lty=2) # incompetitiva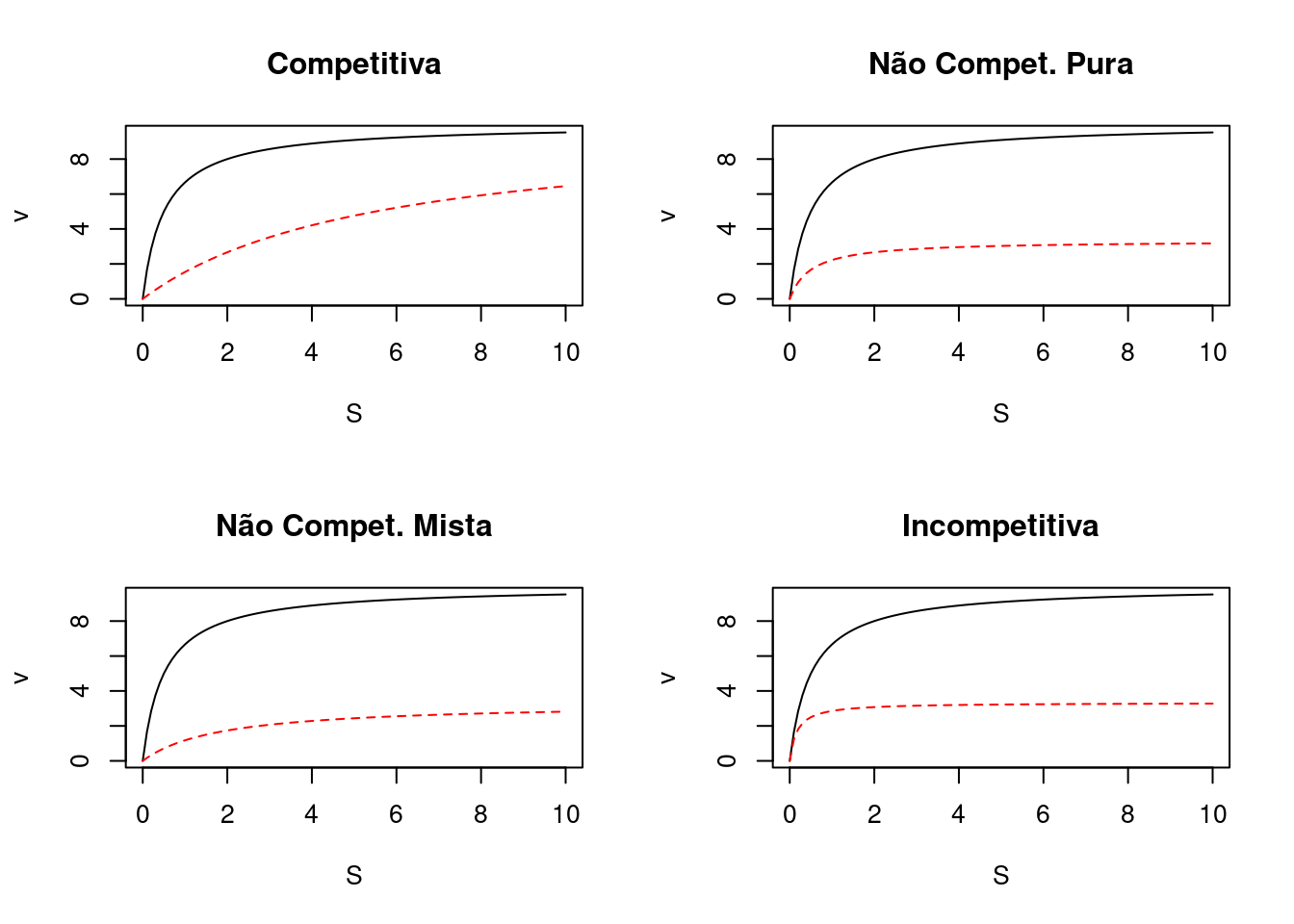
Figura 5.16: Curva de Michaelis-Mentem na presença de inibidores de comportamento clássico.
layout(1) # retorna à janela gráfica originalAinda que seja possível um discernimento do modelo competitivo dos demais, perceba também que isso só foi possível por uma simulação que empregou um teor S 20 vezes maior que o valor de Km da reação. Isso nem sempre é possível na prática, como elencado abaixo, já que o emprego de altos teores de S:
- Agrega maior custo financeiro ao ensaio.
- Pode resultar em inibição por excesso de substrato.
- Pode elevar a viscosidade do meio, reduzindo a taxa catalítica.
5.5.2.2 Diagnóstico de modelos de inibição enzimática por Lineweaver-Burk
R junto à transformação de Lineweaver-Burk (ou qualquer outra), como abaixo.# Substrato e Inibidor
S=seq(0.1,10,length=10) # cria um vetor para substrato
I = 2 # concentração de inibidor
# Parâmetros cinéticos:
Km=0.5
Vm=10
Kic=0.2
Ki=0.2
Kiu=1
# Equações
v=Vm*S/(Km+S) # equação de MM
v.comp=Vm*S/(Km*(1+I/Kic)+S) # competitivo
v.puro=Vm*S/(Km*(1+I/Ki)+S*(1+I/Ki)) # não competitivo puro
v.misto=Vm*S/(Km*(1+I/Kic)+S*(1+I/Kiu)) # não competitivo misto
v.incomp=Vm*S/(Km+S*(1+I/Kiu))
# Gráficos
par(mfrow=c(2,2)) # área de plot pra 4 gráficos
plot(1/S,1/v,type="l",main="Competitivo",ylim=c(0,2))
points(1/S,1/v.comp, type="l",col="red")
plot(1/S,1/v,type="l",main="Puro",ylim=c(0,5))
points(1/S,1/v.puro, type="l",col="red")
plot(1/S,1/v,type="l",main="Misto",ylim=c(0,2))
points(1/S,1/v.misto, type="l",col="red")
plot(1/S,1/v,type="l",main="Incompetitivo",ylim=c(0,1))
points(1/S,1/v.incomp, type="l",col="red")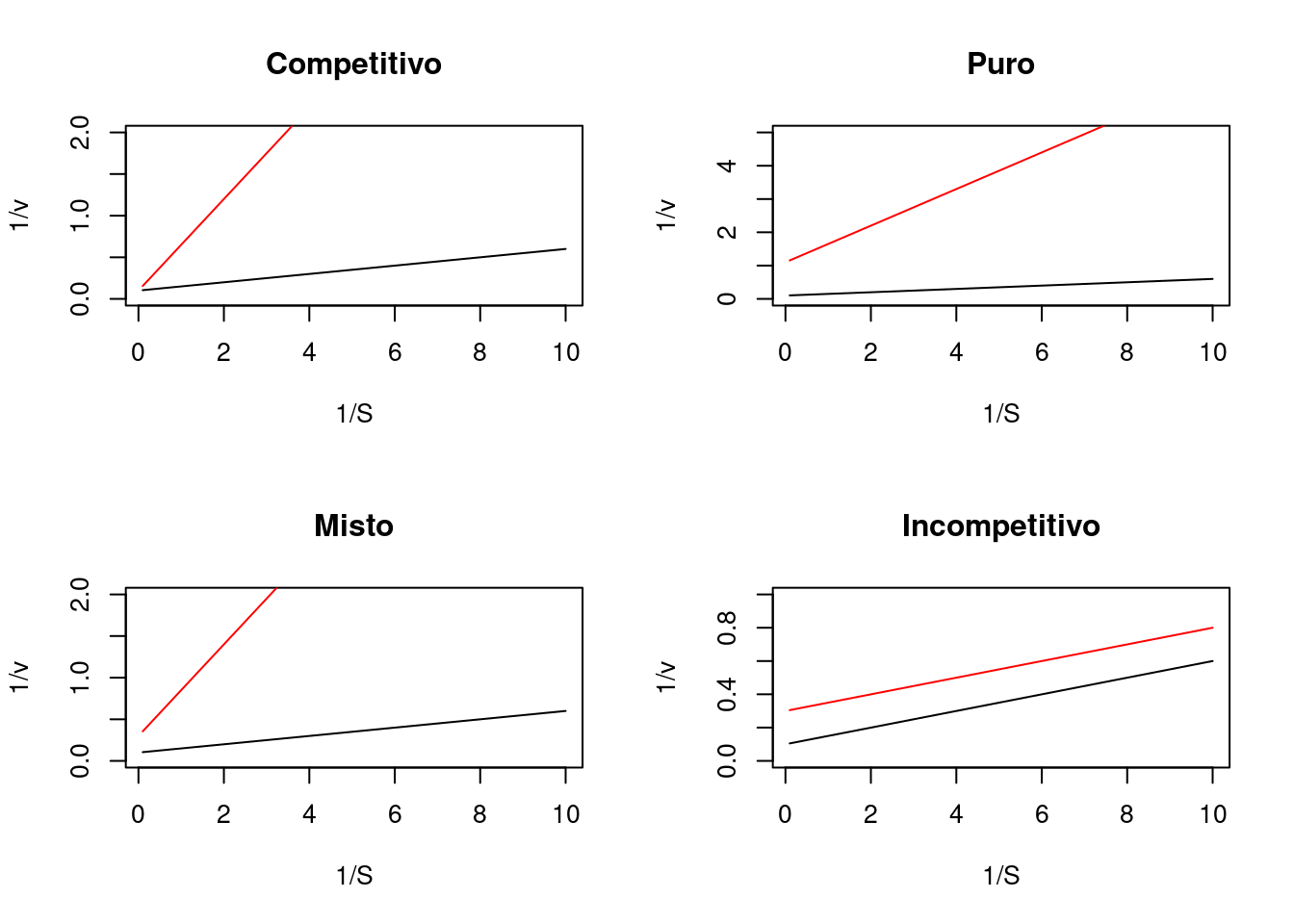
Figura 5.17: Diagnóstico de modelos de inibição enzimática por Lineweaver-Burk.
layout(1) # volta à janela gráfica normalNovamente, ainda que a linearização permita um melhor diagnóstico do tipo de inibição presente, o ajuste não linear é mais adequado para a determinação das constantes de inibição (Ki’s), uma vez que não agrega os erros advindos das transformações lineares (embora a inserção de pesos estatísticos possa aliviar a imprecisão dos resultados).
5.5.2.3 Ki & IC\(_{50}\)
No entanto, existe uma relação útil entre a constante de equilíbrio de dissociação do inibidor Ki e o valor de IC\(_{50}\) que permite sua permuta, desde que conhecido o modelo de inibição (Yung-Chi and Prusoff 1973). Generalizando para os modelos de inibição, pode-se definir uma equação geral pra relação de Cheng-Prusoff como:
R a obtenção de IC\(_{50}\), utilizando-se um ajuste não linear para a equação de quatro parâmetros que segue (curva de Rodbard, DeLean, Munson, and Rodbard (1978)).logI.nM <- c(5.5,5.2,4.9,4.6,4.3,3.7,3.3,3,2.8) # conc. de I, em unidade log10
ativ.res <- c(0.02,0.07,0.12,0.22,0.36,0.53,0.67,0.83,0.85) # ativ. residual, v/Vm
dados <- data.frame(logI.nM,ativ.res) # criação do dataframe
plot(ativ.res~logI.nM, dados) # plot dos dados
ic50.fit <- nls(formula(ativ.res ~ inf+(sup-inf)/(1+(logI.nM/logIC50)^nH)),algorithm="port", data=dados,start=list(inf=0, sup=0.80, logIC50=4,nH=10), lower=c(inf=-Inf, sup=-Inf, logIC50=0, nH=-Inf) ) # ajuste não linear
summary(ic50.fit) # sumário do ajuste##
## Formula: ativ.res ~ inf + (sup - inf)/(1 + (logI.nM/logIC50)^nH)
##
## Parameters:
## Estimate Std. Error t value Pr(>|t|)
## inf -0.321 0.293 -1.09 0.3235
## sup 1.120 0.231 4.85 0.0047 **
## logIC50 4.081 0.231 17.67 1.1e-05 ***
## nH 4.054 1.746 2.32 0.0679 .
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.03 on 5 degrees of freedom
##
## Algorithm "port", convergence message: relative convergence (4)lines(logI.nM,fitted(ic50.fit), col="blue") # linha ajustada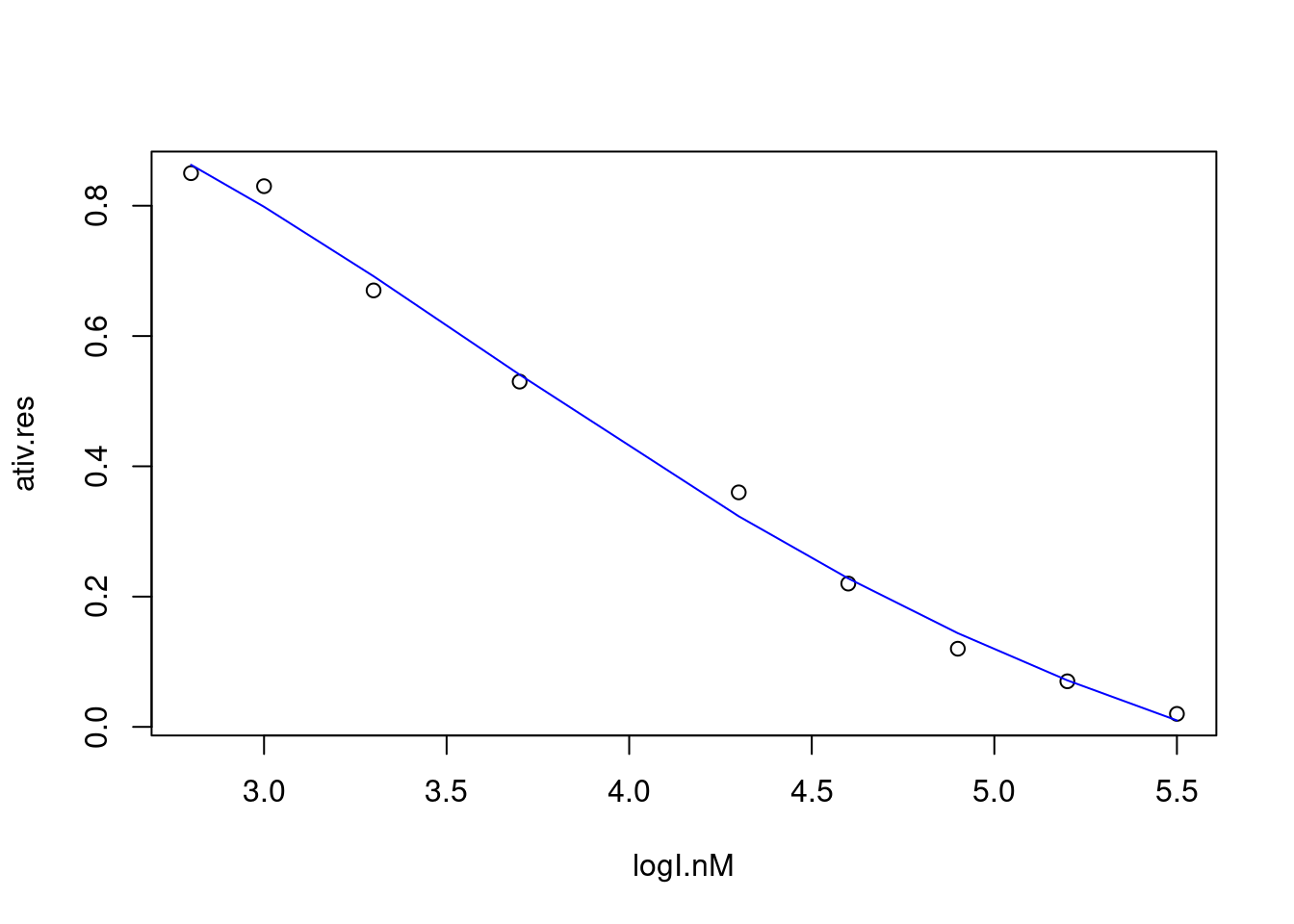
# E para extrair o valor de IC50...
IC50<-10^(coef(ic50.fit)[3]) # extração do 3o. parâmetro da tabela de ajuste, isto é: logIC50:
IC50## logIC50
## 12042coef. Isto é muito útil quando desejamos utilizar um coeficiente obtido em cálculos automáticos (programáveis), como veremos mais adiante. Por ora, faz-se interessante apresentar o parâmetro de IC50 obtido de forma mais elegante.Para isso, podemos utilizar duas funções do
R para exprimir resultados quantitativos junto à caracteres (palavras, frases): print() e cat. O trecho de código abaixo ilustra esse output, e algumas diferenças.cat("Valor de IC50 (nM):", IC50, "\n")## Valor de IC50 (nM): 12042print(paste("Valor de IC50 (nM):", IC50))## [1] "Valor de IC50 (nM): 12042.0414863972"print exibe aspas e indexa o nome da coluna, enquanto cat os omite. Em adição, pode-se perceber outra variação no formato de impressão entre os dois comandos pelo exemplo abaixo:print(paste("teores:",c(10,25,50)))## [1] "teores: 10" "teores: 25" "teores: 50"cat("teores:",c(10,25,50))## teores: 10 25 50R é a de se reduzir o número de casas decimais apresentados. Nesse caso, pode-se utilizar o comando round.IC50<-10^(coef(ic50.fit)[3])
print(paste("Valor de IC50 (nM):", round(IC50,digits=2))) # arredondamento para duas casas decimais## [1] "Valor de IC50 (nM): 12042.04"R para o cálculo de IC50, entre esse o pacote drc (dose-response curve).5.6 Diagnóstico estatístico de inibição enzimática
BIC ou da função AIC do R, e que respectivamente calculam valores para o Critério de Informação Bayseiano (Spiess and Neumeyer 2010) ou do Critério de Informação de Akaike (Akaike 1974). Em comum esse parâmetros calculam um valor relativo de informação não computada por um modelo avaliado. O menor valor encontrado para ambos espelha a solução do melhor modelo de ajuste.Matematicamente, BIC e AIC podem ser expressos como:
|Onde p representa o no. de parâmetros do modelo, n o número total de pontos experimentais, k o fator p+1, e RSE o valor da soma dos quadrados dos resíduos (residual sum squares).
nlstools, provendo o ajuste, plotagem, inspeção de resíduos, e aplicação de BIC e AIC:library(nlstools)##
## 'nlstools' has been loaded.## IMPORTANT NOTICE: Most nonlinear regression models and data set examples## related to predictive microbiolgy have been moved to the package 'nlsMicrobio'comp <- nls(compet_mich, vmkmki, list(Km=1,Vmax=20,Ki=0.5)) # ajuste competitivo, com dados, equação e sementes fornecidas pelo pacote nlstools
plotfit(comp, variable=1) # comando de plotagem do pacote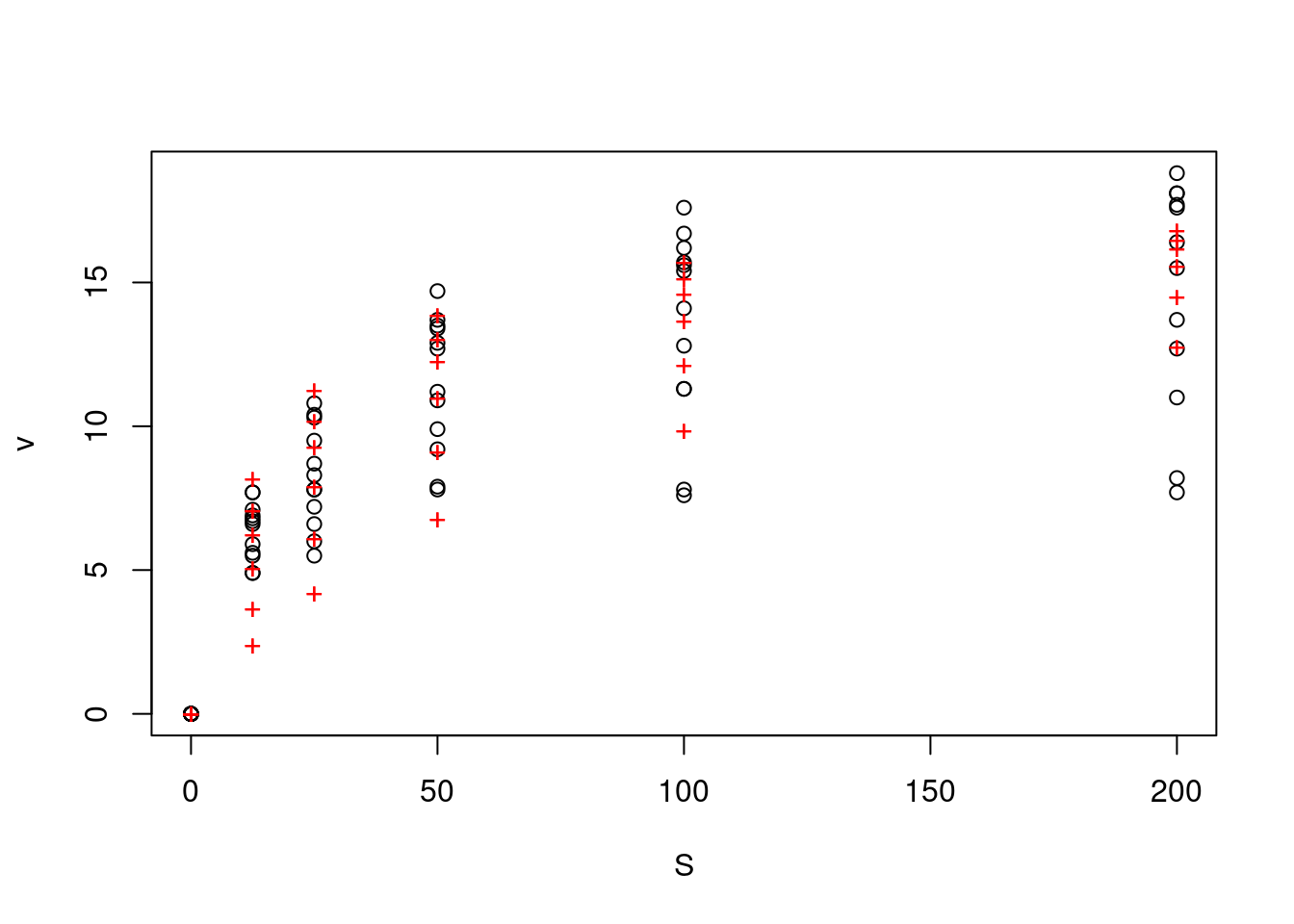
summary(comp)##
## Formula: v ~ S/(S + Km * (1 + I/Ki)) * Vmax
##
## Parameters:
## Estimate Std. Error t value Pr(>|t|)
## Km 15.214 2.501 6.08 5.8e-08 ***
## Vmax 18.056 0.629 28.71 < 2e-16 ***
## Ki 22.282 4.906 4.54 2.3e-05 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 2 on 69 degrees of freedom
##
## Number of iterations to convergence: 11
## Achieved convergence tolerance: 5.11e-06res_comp <- nlsResiduals(comp) # resíduos do ajuste
plot(res_comp, which=1) # plotagem de resíduos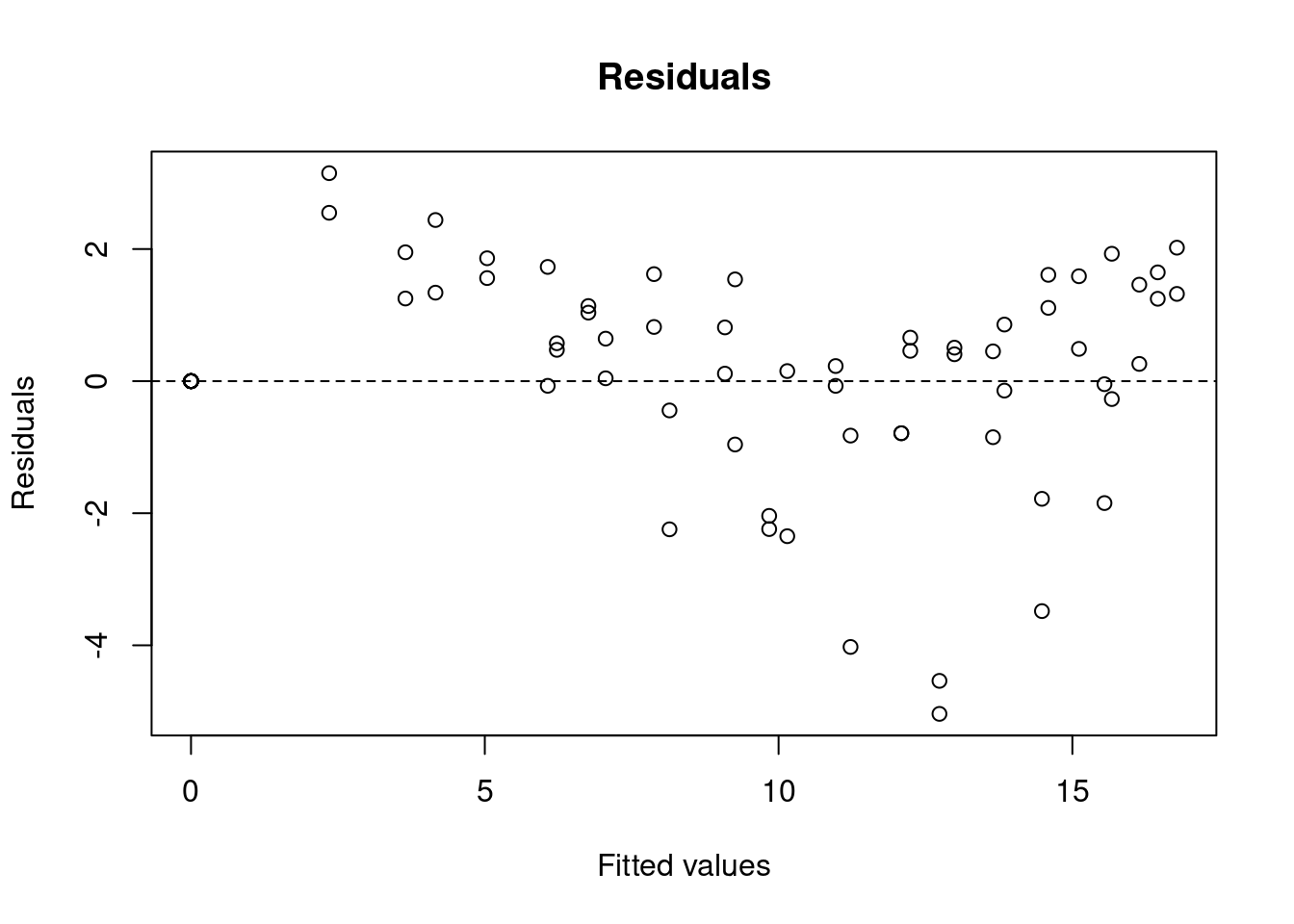
noncomp <- nls(non_compet_mich, vmkmki, list(Km=1, Vmax=20, Ki=0.5)) # o mesmo que acima, mas para o modelo não competitivo
plotfit(noncomp, variable=1)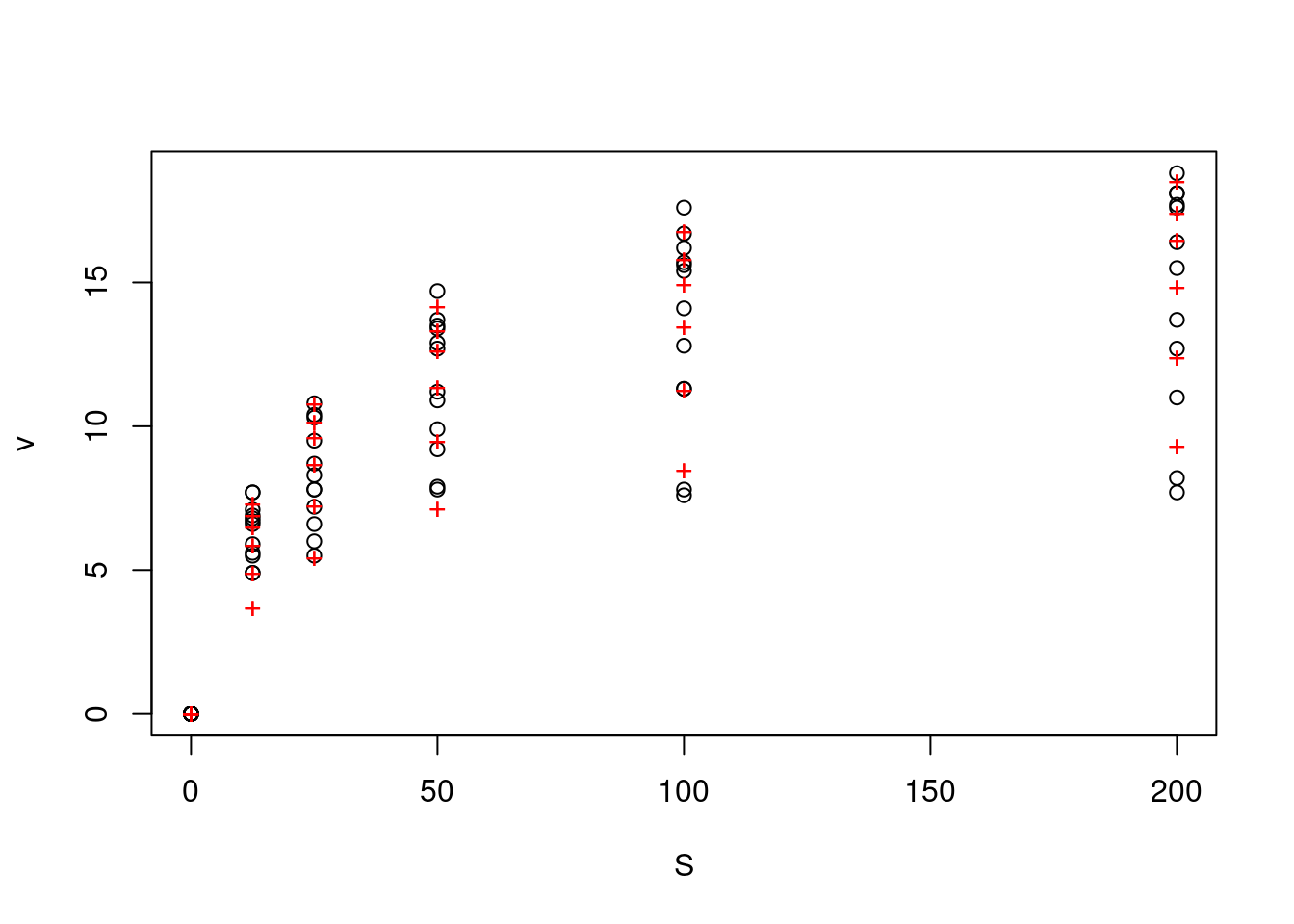
summary(noncomp)##
## Formula: v ~ S/((S + Km) * (1 + I/Ki)) * Vmax
##
## Parameters:
## Estimate Std. Error t value Pr(>|t|)
## Km 22.779 1.474 15.5 <2e-16 ***
## Vmax 20.587 0.431 47.8 <2e-16 ***
## Ki 101.356 7.330 13.8 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.9 on 69 degrees of freedom
##
## Number of iterations to convergence: 7
## Achieved convergence tolerance: 8.25e-06res_noncomp <- nlsResiduals(noncomp)
plot(res_noncomp, which=1)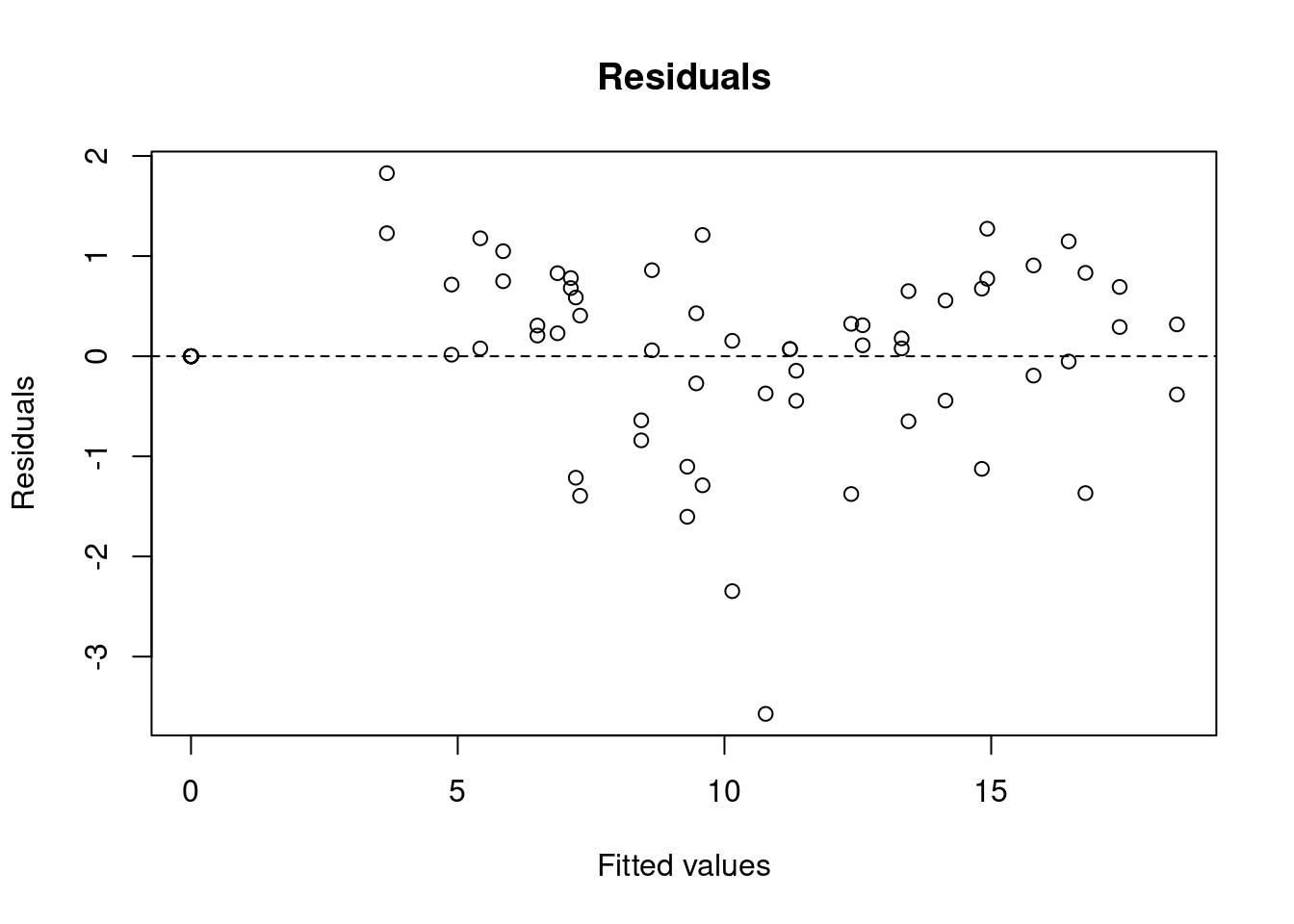
BIC(comp,noncomp) # Critério de informação de Baysean## df BIC
## comp 4 286
## noncomp 4 202AIC(comp,noncomp) # Critério de informação de Akaike## df AIC
## comp 4 277
## noncomp 4 1935.7 Cinética de estado pré-estacionário
A cinética de estado pré-estacionário segue um formalismo um pouco distinto, e que depende do quantitativo de etapas reacionais. Exemplificando abaixo para uma reação de 3 etapas (Johnson 1992):
Onde kobs e Ao representam parâmetros experimentais de constante de velocidade observada e amplitude, respectivamente. Esses parâmetros podem ser obtidos a partir do ajuste não linear da equação abaixo aos dados experimentais:
\[\begin{equation} P=Ao(1-e^ {-kobs} + kcat * t) \tag{5.21} \end{equation}\]# Parâmetros
k2=387
km2=3
k3=22
xmin=0
xmax=0.075 # definição de limites para função
# Variáveis da equação de simulação (função dos parâmetros)
kobs=k2+km2+k3
Ao=k2*(k2+km2)/kobs^2
kcat=k2*k3/kobs
# Definição da função de simulação
sim=function(x,kobs,Ao,kcat){Ao*(1-exp(-kobs*x))+kcat*x}
# Curval de simulação
curve(sim(x,kobs=kobs,Ao=Ao,kcat=kcat),col="blue",
type="o",xlim=c(xmin,xmax),cex=0.5,
xlab="tempo", ylab="[P]")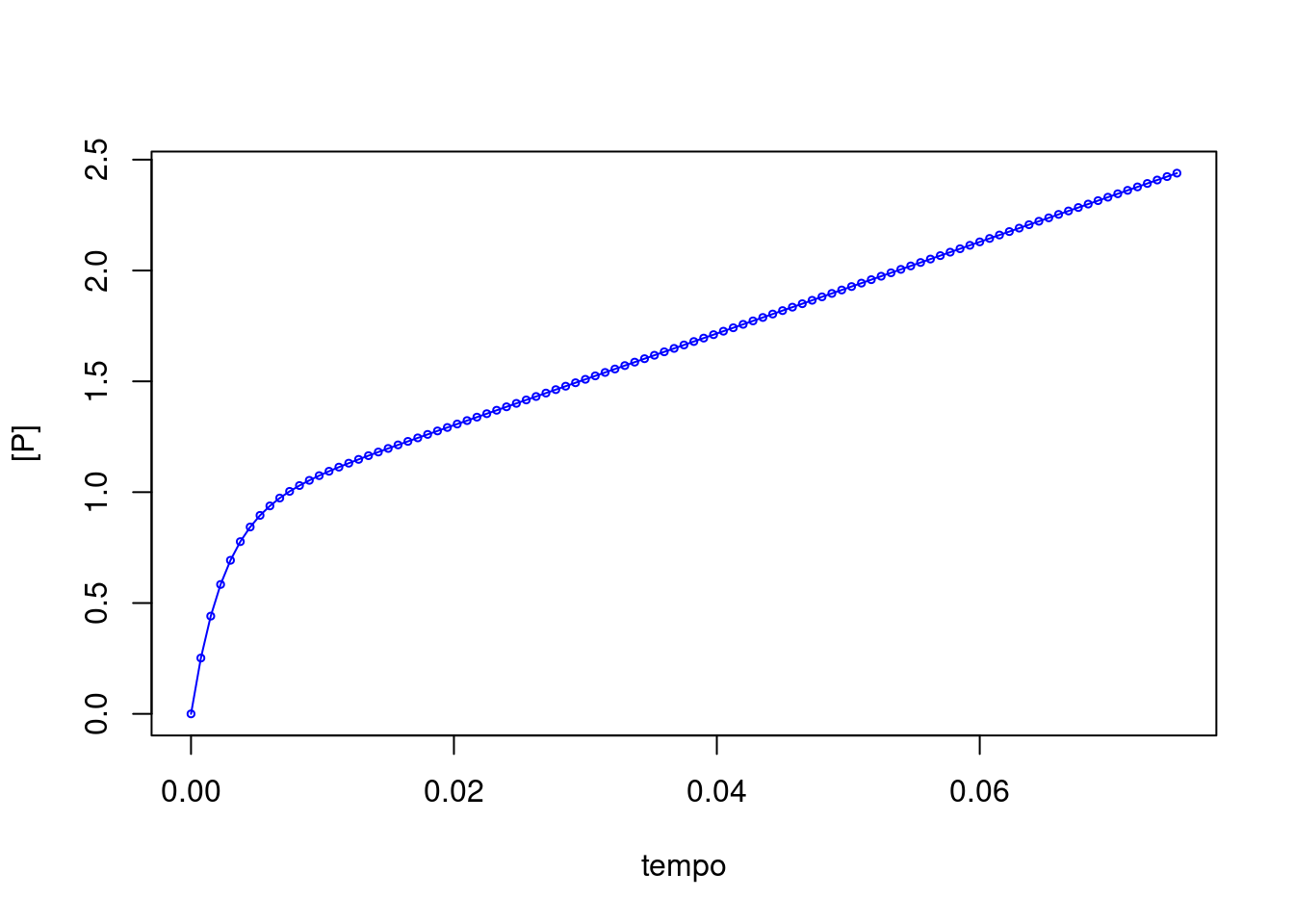
Figura 5.18: Formação de produto num modelo cinético de estado pré-estacionário.
R possui funções de minimização que permitem encontrar a raíz de equações lineares ou não lineares.O procedimento envolve minimizar iterativamente um vetor de equações dadas as sementes para cada parâmetro. Para tal busca-se obter f(x) = 0 pela diferença entre um valor de referência; ou seja, quando a solução encontrar x quando f(x)-y = 0. Exemplificando, supondo que f(x) seja a+b/x, e que y seja 3. Então a busca se dá no sentido de encontrar a e b em a+b/x-3.
Para a determinação das constantes de velocidade representadas na cinética transiente, vale mencionar a função
optim em stats ou o pacote rootSolve, que buscam minimizar equações lineares e não lineares para encontrar os valores de seus parâmetros.Na solução dos parâmetros para estado pré-estacionário, ilustra-se abaixo o emprego do
R com rootSolve, adicionando ainda a busca para Km como segue.# Cálculo de constantes cinéticas por solução de sistema de equações não lineares aplicadas à cinética de burst.
library(rootSolve)
kobs = 0.06; Ao = 50; kcat = 300; Ks = 15
# define os parâmetros de ajuste não linear obtidos por curva progressiva experimental, t x P;
# Obs: Ks obtido experimentalmente de curva de S x kobs
# Parâmetros
# x[1]=k2
# x[2]= k3
# x[3] = Km
# Modelo
model = function(x) c(x[1]/kobs^2-Ao,(x[1]*x[2])/kobs-kcat,Ks*x[2]/(x[1]+x[2])-x[3])
# o modelo acima deve conter uma lista de equações cuja igualdade é zero, ou seja, f(x)=0
(ss=multiroot(model,c(1,1,1))) # comando de execução do rootSolve (sementes pro algoritmo)## $root
## [1] 0.2 100.0 15.0
##
## $f.root
## [1] 0e+00 1e-13 -1e-09
##
## $iter
## [1] 4
##
## $estim.precis
## [1] 4e-10- root = valores de xi pra f(xi)=0 ; ou seja, k2, k3, e Km;
- f.root = valor de cada função pra cada xi (deve ser próximo de zero para cada);
- iter = no. iterações ;
- esti.precis = estimativa da precisão.
R na solução de problemas quantitativos em biofísico-química. Dessa forma, omitimos diversos conceitos, tais como cinética lenta de interação de substrato (slow binding), cinética de múltiplos substratos (reação sequencial e ping-pong), equação integrada de Michaelis-Menten e curvas progressivas, ativação de moduladores, influência de pH e temperatura na catálise, e enzimas multisítios, entre vários.